Organelas Membranosas
-
Upload
matheus-correa -
Category
Documents
-
view
15 -
download
1
description
Transcript of Organelas Membranosas

ORGANELAS MEMBRANOSAS
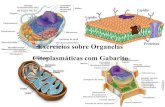
As células eucarióticas apresentam compartimentos isolados por membranas dentro da célula, as organelas. Essas organelas fazem uso de diversas proteínas, para selecionar a entrada de moléculas na organela, para catalisar as reações que nela ocorrem, para emitir informações, entre outros.
COMPARTIMENTOS DOS EUCARIOTOS
Os eucariotos apresentam basicamente o mesmo conjunto de organelas
As membranas são vitais para as células. Sem elas, não seria possível realizar a fosforilação oxidativa ou realizar o metabolismo lipídico. Para uma área maior de reação, a célula criou compartimentos especializados, que podem realizar as mais diversas funções da célula.
O núcleo contém o genoma da célula (com excessão do DNA mitocondrial), e é a principal área de produção de RNA e DNA. O citoplasma é constituído do citosol e das organelas. O citosol é aonde ocorrem a maioria das reações de sinalização e síntese proteica.
O retículo endoplasmático (RE) pode conter ribossomos (Rugoso), ou não (Liso). Os ribossomos a medida que produzem as proteínas, as lançam no RE. Ele ainda é a principal fonte de lipídeos da célula, e armazena Ca++. É muito comum que o RE mande seus produtos para o complexo de Golgi (CG), composto por sacos denominados cisternas, aonde ocorre modificação covalente de muitas moléculas.
A mitocôndria produz a maior parte da energia na célula. Os lisossomos contém enzimas digestivas para a degradação de organelas “mortas”, macromoléculas e partículas do meio externo. O material endocitado precisa passar pelos endossomos antes de chegar ao lisossomo. Os peroxissomos contém enzimas para reações de oxidação.
Para exercer as funções específicas das células, as organelas encontram-se em quantidades diferentes, e podem realizar funções adicionais. Também para melhor desempenhar suas funções, as organelas se posicionam adequadamente, com ajuda do citoesqueleto.
Evolução das organelas
Acredita-se que o precursor das células eucariotas seja uma célula simples, sem organelas, e bem pequena. A membrana plasmática realizava todas as funções dependentes de membrana. Com o crescimento da célula, cresceu também a razão área/volume, impedindo que apenas a membrana plasmática suprisse todo o volume celular. Para desviar desse problema, as células criaram membranas internas para realizar parte das funções membranares.
As organelas intracelulares provavelmente se originaram da própria membrana, que invaginou e se desligou da membrana, formando o RE, CG, lisossomo e endossomos. As

mitocôndrias provavelmente se originaram de bactérias aeróbicas endocitadas, sendo a membrana interna a membrana da bactéria, e a membrana externa, parte da membrana plasmática.
São três grandes grupos de organelas nos humanos: O núcleo e o citoplasma, que se comunicam por poros, logo são contínuos; todas as organelas, com uma membrana apenas e; a mitocôndria.
Movimento de proteínas entre compartimentos
As proteínas são sintetizadas no citosol, com exceção das que são sintetizadas por ribossomos mitocondriais. Seu destino depende de sinais de direcionamento, que as levam para uma outra organela, ou, na sua ausência, as faz permanecer no citosol. Os sinais, como os demais, precisam de receptores de direcionamento, seja qual for o meio de transporte. Esses receptores são relativamente inespecíficos, reconhecendo grupos de proteínas, e após seu uso, ocorre catálise do complexo proteína-receptor, para que o receptor possa ser reutilizado.
Pode ocorrer transporte por poros, entre o citoplasma e o núcleo. Os poros funcionam como portões seletivos, que promovem transporte ativo de macromoléculas, ou transporte passivo de moléculas menores.
Também pode ocorrer transporte transmembranar, por proteínas transmembranares que movimentam proteínas do citosol para as organelas membranosas. Geralmente é necessário que a proteína se desdobre para passar pelo transportador.
A terceira forma é por transporte vesicular, onde proteínas são englobadas por uma membrana, formando uma vesícula. Essa vesícula vai até outra organela, suas membranas se fundem, e a proteína é despejada dentro da organela.
Sequências de sinal
Os sinais de direcionamento podem ser sequências de aminoácidos, as sequências sinalizadoras. Estas podem encontrar-se no terminal da proteína (geralmente o N), e ser removidos por uma peptidase de sinal, após o processo de direcionamento. A sequência pode estar no meio da proteína, permanecendo parte dela, ou pode estar em várias partes, formando uma chave sinal, tridimensional. Cada sequência dá um endereço à proteína, devido principalmente às suas propriedades, e secundariamente à sequência exata de aminoácidos.
Informação intrínseca das organelas
Na divisão celular, as organelas são aumentadas e então divididas. Não se pode retirar uma organela e esperar que ela se recomponha. Cada organela apresenta proteínas específicas na sua membrana, muitas delas sendo produtos da própria organela. Para algumas organelas se formar, além do DNA, precisam de ao menos uma proteína específica para manter o padrão de compartimentos da célula original. Algumas organelas, no entanto, podem se originar de outras, como os endossomos.

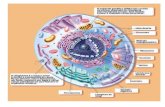
O TRANSPORTE DE MOLÉCULAS ENTRE O CITOSOL E O NÚCLEO
O envelope nuclear delimita o núcleo e o citosol, isolando o DNA. O envelope contém duas membranas, penetradas por poros, e de constituição diferente. A membrana nuclear interna contém proteínas-âncora para a cromatina e para a lâmina, uma rede de proteínas que dá suporte ao núcleo. A membrana nuclear externa é contínua com o RE, e contém ribossomos que lançam seus produtos no espaço perinuclear.
O tráfego é bidirecional. Pode ocorrer importação de material citosólico, e exportação do material produzido no núcleo. Não exclusivamente, os dois processos podem ocorrer numa molécula. Uma proteína ribossomal é formada no citoplasma, vai para o núcleo e recebe rRNA, voltando ao citosol.
Os complexos porosos
Atravessando o envelope nuclear estão os nuclear pore complexes (NPCs), ou complexos de poros nucleares. Os NPCs são constituídos de nucleoporinas ou proteínas NPC, que se arranjam e se repetem de forma extremamente complexa. Os NPCs conseguem trabalhar com uma velocidade muito grande, e nos dois sentidos ao mesmo tempo. Os poros permitem a passagem livre de moléculas hidrofílicas pequenas, mas acredita-se que algumas das proteínas do complexo não apresentam estruturas organizadas, ficando embaralhadas no meio do poro, impedindo a passagem de moléculas grandes. Dessa forma, ainda que sejam compartimentos diretamente ligados, o citosol e o núcleo podem manter diferentes tipos de proteínas.
Para a passagem de moléculas grandes, existem receptores específicos.
Sinais de localização nuclear
As proteínas nucleares apresentam uma boa capacidade de retornar ao núcleo, pois contém os sinais de localização nuclear, sequências pequenas de aminoácidos específicas, localizadas em vários pontos da proteína.
Durante a passagem de moléculas grandes, os poros se abrem de forma ativa. Duas fibrilas em forma de tentáculo pegam as moléculas e as passam pelo poro, onde as proteínas que bloqueavam a passagem foram afastadas. A molécula que passa pelo poro é comprimida, pois sofre reestruturação durante o processo.
Receptores de importação nuclear
Para entrar no núcleo, os sinais de localização nuclear precisam ser reconhecidos pelos receptores de importação nuclear. Eles são proteínas citosólicas solúveis, que se ligam ao sinal de localização e às nucleoporinas, entre elas, as que formam fibrilas citosólicas. As fibrilas são compostas por proteínas com FG-repeats, repetições extensas de sequências com fenilalanina e glicina, que servem como doca para os receptores de importação. Acredita-se que todo o caminho dentro do poro, todas as partes das proteínas NPC pelas quais os receptores e as proteínas carregadas passam são cobertas de FG-repeats. Acredita-se que o receptor vai se ligando, dissociando, e ligando novamente, pulando de FG em FG, de forma que passe pelo

emaranhado dentro do poro. No fim, a carga e o receptor se dissociam, e o segundo volta ao citosol.
Nem todas as vezes a proteína carregada se liga direto ao receptor. Existem casos em que uma proteína auxiliar, um adaptador, forma uma ponte entre o sinal de localização nuclear e o receptor de importação nuclear.
Exportação nuclear
Para a exportação, se usam os sinais de exportação nuclear, e os receptores de exportação nuclear. Os receptores se ligam às nucleoporinas e às proteínas com sinais de exportação.
Os receptores de importação e exportação são relacionados, e são decodificados por uma família de genes, a família dos receptores de transporte nuclear, ou carioferinas. O funcionamento dos exportadores é o inverso dos importadores.
Ran GTPase
A importação aumenta o nível de organização celular, processo que não é espontâneo. A energia é adquirida do GTP hidrolisado pela enzima Ran GTPase, presente no núcleo e citosol, essencial para a importação e exportação.
Como outras GTPases, a Ran pode estar na conformação GTP ou GDP. Duas proteínas regulatórias da Ran controlam a conversão. A GAP (GTPase-activating protein) ativa a Ran-GTP, hidrolisando o GTP e mudando-a para Ran-GDP. A GEF (Guanine exchange factor) troca o GDP por GTP. A Ran-GAP é citosólica, e a Ran-GEF é nuclear, de forma que o citosol contenha Ran-GDP, e o núcleo Ran-GTP.
Essa organização permite que haja uma direção no transporte por nucleoporinas. Os importadores se ligam às FG-repeats e saem pulando. Ao chegar no núcleo, a Ran-GTP se liga ao importador, despejando a carga (não é necessário que haja carga para os importadores chegarem ao núcleo). Como o Ran-GDP não se liga aos importadores, não ocorre descarga no citosol. Com o Ran-GTP, o importador volta ao lado citosólico, aonde as Ran-GAPs transformam a Ran-GTP em Ran-GDP.
O mesmo ocorre nos exportadores, só que o Ran-GTP promove a ligação da proteína carregada. Dessa forma, o exportador vai ao lado citosólico com a proteína e com a Ran-GTP. Chegando no citosol, as Ran-GAPs promovem a hidrólise do GTP, e a descarga. O exportador volta ao lado nuclear.
Controle de acesso
Algumas proteínas, como as ribossomais, contém sinais para importação e exportação, de forma que fiquem entrando e saindo continuamente do núcleo, formando um equilíbrio. Deslocando-o, a proteína se concentra no citosol ou no núcleo. Em outros casos, a célula pode prevenir que proteínas regulatórias de genes entrem no núcleo, ou podem (des)fosforilar os sinais de importação e exportação.

Algumas proteínas regulatórias de genes são presas no citosol por proteínas regulatórias, que as ancoram ou disfarçam seus sinais de localização nuclear.
A exportação de RNA também tem regulação. Proteínas específicas se ligam ao RNA durante a transcrição e separação, e ao chegar no citosol são separadas e voltam ao núcleo.
Carioteca na mitose
A lâmina nuclear é uma rede de filamentos intermediários formados pelas laminas. A lâmina fornece forma e suporte à carioteca, e fica presa às proteínas NPC e a proteínas integrais da membrana interna. Elas interagem com a cromatina, que interage com outras proteínas da membrana.
Durante a mitose, a quinase Cdk promove a degradação da lâmina, e ocorre também por fosforilação a degradação da membrana interna. As nucleoporinas ficam dispersas, algumas ligadas aos importadores. O Ran-GEF e GAP promovem o posicionamento da cromatina na célula nova, pois o GEF fica ligado à cromatina. A Ran-GTP se acumula perto do DNA, promovendo a dissociação das nucleoporinas dos importadores, e formação de novos NPCs. Fora isso, as proteínas integrais e as laminas se ligam de novo à cromatina. As membranas do RE envolvem a cromatina e formam uma nova carioteca.
Durante o processo, apenas as proteínas ligadas à cromatina ficam no núcleo, as demais precisam ser importadas novamente.
Diferentemente das outras organelas, as proteínas do núcleo não perdem seus sinais de localização, pois precisam ser constantemente importadas. As moléculas de outras organelas ficam nela de geração em geração, após serem importadas uma vez.
MITOCÔNDRIAS
A mitocôndria produz ATP pela fosforilação oxidativa. Ela contém seu próprio mecanismo para síntese de proteínas, mas boa parte delas é produzida no núcleo. Ela apresenta duas membranas. A interna delimita a matriz, e forma extensas invaginações chamadas cristas. A externa delimita o espaço intermembranar e está em contato com o citosol. A divisão da mitocôndria requer crescimento, dependente da importação de proteínas, e fissão. A entrada de proteínas geralmente é chamada de translocação de proteínas.
Sequências sinais e translocadores
As proteínas são produzidas e liberadas no citosol como proteínas mitocondriais precursoras, e são colocadas na mitocôndria após a tradução. Essas proteínas apresentam sequências peptídicas específicas. Muitas proteínas da matriz têm o sinal no terminal N, que é prontamente removido após a entrada. Outras, como todas as da membrana externa, e muitas do espaço IM e membrana interna têm sequências não removíveis. As sequências das proteínas matriciais contém uma α-hélice com ponta positiva e polar, e base apolar.

Os translocadores são complexos proteicos. O TOM faz o transporte pela membrana externa, e os TIMs pela interna. Eles têm domínios que agem como receptores para as proteínas precursoras, e outras que funcionam como canais de translocação.
O TOM é necessário para a passagem de todas as proteínas feitas no núcleo para a mitocôndria. Ele coloca a sequência-sinal no espaço IM e ajuda a inserir proteínas transmembranares na membrana externa. Aí entra o complexo SAM, que ajuda a dobrar a proteína. O complexo TIM23 transloca proteínas da matriz e ajuda a inserir proteínas na membrana interna. A TIM22 promove a adição de proteínas na membrana interna, e o complexo OXA promove a inserção de proteínas na membrana interna que foram produzidas na mitocôndria. O OXA também ajuda a inserir proteínas na membrana interna que vieram de outros translocadores.
Importação dos precursores
Os precursores não são sintetizados na sua conformação correta. Eles ficam presos a proteínas citosólicas, como as chaperonas da família Hsp70, generalizadas, ou proteínas específicas para o sinal localizador. Ao chegar no complexo TOM, o receptor reconhece o sinal localizador e promove a passagem da sequência-sinal e desassociação de proteínas auxiliares.
A passagem das proteínas precursoras acontece diretamente pelas duas membranas, sem precisar ser totalmente injetada no espaço IM para chegar à matriz. A sequência-sinal é inserida pelo complexo TOM num complexo TIM23, ativando-o e passando a proteína direto para a matriz. Ainda assim, eles podem agir independentemente.
Recursos energéticos para a importação
A mitocôndria requer hidrólise de ATP para fazer a importação, e um gradiente de membrana.
A primeira necessidade é na ligação do precursor com o TOM, pois as chaperonas da família Hsp70 requerem hidrólise de ATP para se dissociar da proteína precursora.
A segunda necessidade é a de um gradiente elétrico, para movimentar a ponta positiva para dentro da matriz. A presença de H+ no espaço IM dá uma carga negativa na matriz, facilitando o movimento da proteína.
A terceira necessidade é na hora de colocar a proteína para dentro da matriz. Com a sequência-sinal inserida, a proteína se liga à uma chaperona Hsp70 mitocondrial, que funciona como um motor, pois é capaz de mudar de conformação com hidrólise de ATP.
Ainda é necessário ATP para dobrar a proteína, pois ela é auxiliada pelas chaperonas mitocôndrias Hsp60, num ciclo de ligação e desassociação.
Inserção de porinas
As porinas formam β-barris que permitem a passagem de água, íons inorgânicos e metabólitos. Elas são inseridas no espaço IM pela TOM, depois são associadas à chaperonas,

para evitar que se enovelem, e depois são inseridas na SAM, que promove a formação do barril.
Transporte na membrana interna e espaço IM
No processo de inserção na membrana interna requer uso da TOM e da TIM23. Passando a sequência-sinal pela TIM23, uma outra sequência hidrofóbica, chamada stop-transfer sequence, ou sequência de parada de transferência, impede que a TIM23 continue a mandar a proteína para a matriz, enquanto a TOM coloca o resto da proteína no espaço IM. A sequência-sinal é clivada, e a proteína permanece ancorada na membrana IM pela sequência de parada.
Outra via é a passagem da proteína para a matriz, clivagem da sequência-sinal, revelando a sequência hidrofóbica, que guia a proteína até a OXA, que a insere na membrana interna.
Algumas delas permanecem ancoradas à membrana, mas algumas são clivadas por proteases, liberando-as no espaço IM. Algumas funcionam como proteínas periféricas de complexos com proteínas transmembranares.
Os transportadores de metabólitos pequenos também podem ser inseridos pela TOM22, sem sinais removíveis no terminal, com sinais no meio da sequência de aminoácidos. Os transportadores são proteínas transmembranares multipasse, com regiões hidrofóbicas aonde a proteína está em contato com a membrana. Não se usam as chaperonas nessa via.
PEROXISSOMOS
São organelas utilizadas para a oxidação. Conseguem proteínas da importação citosólica ou via RE. Essas reações eram usadas por bactérias que sofriam com a toxicidade do O2. Hoje, nas células humanas, realizam funções que não foram tomadas pela mitocôndria.
Reações de oxidação
Os peroxissomos usam o O2 para retirar hidrogênio de moléculas orgânicas, formando peróxido de hidrogênio:
RH2 + O2 R + H2O2
O peróxido de hidrogênio é usado para desintoxicar várias moléculas no sangue, transformando-as em água, na peroxidação catalisada pela catalase:
R’H2 + H2O2 R’ + 2H2O
O peróxido em excesso é transformado em água:
2H2O2 2H2O + O2

Eles também podem promover a β oxidação de ácidos graxos, removendo grupos de dois carbonos da cadeia alquila do ácido graxo, transportando o acetil-CoA gerado para o citosol, para uso em vias biossintéticas.
Eles também catalisam as reações iniciais na formação dos plasmalogênios, os lipídeos mais abundantes na mielina, sendo, portanto, essenciais para o sistema nervoso.
Importação
São dependentes de sequências-sinal, como os demais. Utilizam-se proteínas receptoras no citosol, que as ancoram na face citosolica da membrana do peroxissomo. As peroxinas são as proteínas que participam da importação. Um complexo com 6 proteínas forma um translocador que não requer desdobramento proteico, e uma proteína Pex5 tem um papel semelhante ao dos importadores nucleicos.
Replicação
Acredita-se que novos peroxissomos se originam de crescimento e fissão, mas também do RE. Proteínas de membrana dos peroxissomos podem ser feitas no citosol, mas algumas se ligam no RE, que forma uma vesícula precursora de peroxissomos, que realiza a importação de proteínas citosólicas e cresce num peroxissomo maduro.
RETÍCULO ENDOPLASMÁTICO
É uma organela formada por túbulos ramificados e sáculos interconectados, contínuos com a membrana externa do núcleo. Ela é vital na síntese proteica e lipídica da célula. O RE sintetiza todas as proteínas transmembranas e lipídeos de várias organelas, e muitas proteínas citosólicas e do próprio lúmen do RE.
Diversidade
O RE varia de função de acordo com a especificidade da célula e com a área do próprio RE. O processo de transporte, diferente das demais organelas, é co-translacional, pois ocorre enquanto o ribossomo produz a proteína. Síntese proteica é uma função do RER.
O REL é importante na síntese de lipídeos e formação de vesículas. Essas partes que formam vesículas são chamadas de retículo endoplasmático transicional. Nos hepatócitos o REL produz lipoproteínas e contém enzimas de desintoxicação.
Além disso, especialmente nos músculos (retículos sarcoplasmáticos), o RE tem a função de armazenar Ca2+, e a potência é proporcional ao número de proteínas transmembranares que se ligam ao Ca2+.
Sinalização
A grande maioria das proteínas tem que passar pelo RE, independente do seu destino. Portanto, além do sinal de importação para a organela definida, elas ainda recebem outro sinal para a importação do RE, o sinal líder, que é clivado por uma peptidase.

Signal-Recognition Particle
Os sinais do RE são levado ao RE por duas moléculas, a SRP, que vai do citosol ao RE e se ligam ao sinal, e o receptor da SRP, na membrana do RE. O SRP é uma molécula complexa, com seis cadeias polipeptídicas e uma molécula de RNA. O SRP é encontrado em todas as células.
O SRP é uma estrutura em forma de vara que envolve o ribossomo. Um fim do SRP se liga à sequência-sinal da proteína em síntese, e o outro, ao sítio de ligação do fator de alongamento no ribossomo. Esse bloqueio impede a produção da proteína. Essa pausa permite que o ribossomo se ligue ao RE, de forma que a proteína seja produzida para dentro do RE. Isso é importante para a produção de enzimas de lisossomos, que podem destruir a célula. Além disso, essas células que produzem muitas hidrolases também apresentam altas concentrações de inibidores. Fora isso, a ligação ao RE impede que a proteína se dobre, sem a necessidade de chaperonas para manter a proteína desdobrada.
O SRP e o ribossomo se ligam ao receptor do SRP, proteína transmembrana, que traz esse complexo para próximo de um translocador. O SRP e o receptor se soltam e o ribossomo pode produzir a proteína desdobrada.
A ligação com SRP cria duas populações de ribossomos, ligados à membrana ou livres, que produzem proteínas diferentes durante um determinado momento. Vários ribossomos podem se associar a uma molécula de mRNA, formando um polirribossomo. Eles são atraídos pelas sequências-sinal de polipeptídeos em crescimento. Ao terminar a síntese, o mRNA fica ligado a membrana, mas o ribossomo pode voltar a ser livre.
Poro aquoso no translocador
As proteínas produzidas nos ribossomos ligados ao RE passam por um poro aquoso numa unidade translocadora, o complexo Sec61. O poro permanece fechado até que ocorra a translocação de proteínas, para evitar que íons inorgânicos, como o Ca2+, escapem. A Sec61 apresenta uma abertura no seu lado, aonde a sequência-sinal pode ser clivada e despejada na membrana (lembrar que ela é hidrofóbica), sem falar que a abertura também ajuda a integrar proteínas na membrana. O ribossomo se associa ao complexo, onde o espaço dentro do ribossomo permanece contínuo ao lúmen do RE.
Translocação pós-translação no RE
É um processo mais comum em bactérias e leveduras. Nos eucariotos o mecanismo utilizado é o da BiP. Um grupo de proteínas acessórias se associa ao complexo Sec61, depositando chaperonas tipo Hsp70 (a BiP, Binding Protein), que funciona, assim como as Hsp70 na mitocôndria, como um pequeno motor. Essas proteínas que vão ao RE pós-tradução são produzidas no citosol e ligadas a chaperonas, para não se dobrarem.
Proteínas transmembranares de uma passagem
Após ser liberado pelo SRP, e a cadeia começa a crescer o suficiente, o poro é aberto pela sequência-sinal do RE, ou start-transfer signal, que se liga a um sítio ativo do mesmo. O

reconhecimento duplo aumenta a segurança do RE, evitando que proteínas indesejadas entrem nele.
O poro apresenta então duas aberturas: o poro aquoso, e um canal lateral. O poro aquoso permite a passagem da cadeia hidrossolúvel, e o canal lateral permite a saída do peptídeo sinalizador que é clivado, e depois é degradado na membrana.
O primeiro caso de inserção de uma proteína TM de uma passagem na membrana do ER é simples. Uma sequência de parada de transferência, stop-transfer signal, para a ação do translocador, onde essa sequência ancora a proteína na membrana. Após a clivagem do peptídeo-sinal, o canal lateral do poro se abre e solta a proteína na membrana.
Nos demais casos, a sequência-sinal do SRP é interna, e ele se liga a ela, levando o ribossomo até a membrana. Lá, o SRP funciona como o start-transfer signal, e começa a translocação da proteína, que vai parar na sequência-sinal do SRP, funcionando a sequência que o encaminhou para o RE, como o stop-transfer signal.
A diferença dos dois casos é se o terminal C ou N fica no lado luminal do RE. Isso depende da carga dos aminoácidos que circundam a sequência sinal. O lado com mais cargas positivas fica no citosol.
Proteínas multipasse
Proteínas que tem mais de uma sequência passando pela membrana são translocadas de formas diferentes. As proteínas de dois passes tem uma sequência de iniciação no meio da proteína, e outra de parada, ficando ancorado por essas duas. Em moléculas mais complexas, após a primeira sequência de iniciação aparece outra sequência de iniciação, e uma de parada, e assim continua até o fim da proteína.
Como já foi dito, a inserção do grupo hidrofóbico na membrana depende dos aminoácidos circundantes, definindo sua orientação. Dessa forma, a orientação das proteínas no RE induz a orientação das proteínas de todas as organelas.
Proteínas residentes
Algumas proteínas não saem do RE, são as proteínas residentes, que apresentam um sinal de retenção. Essas proteínas realizam reações no RE, e ajudam as proteínas transitórias a se dobrar normalmente.
Uma dessas é a proteína dissulfídico isomerase, que oxida grupos SH em ligações S-S. Outra é a BiP, importante na importação proteica e no reconhecimento de proteínas dobradas de forma errada. Ela se liga aos locais de erro e impede a saída dessas proteínas do RE. A BiP utiliza ATP para modificar sua conformação.