La celula (organelas y transcripción)
-
Upload
maria-laura -
Category
Documents
-
view
27 -
download
0
description
Transcript of La celula (organelas y transcripción)

Laura Mendoza~ 1 ~
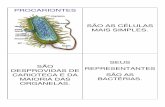
El comportamiento de una célula está determinado no solo por su genoma sino también por cuales de estos genes se expresan en un momento determinado. En esto se basa la diferenciación celular. La regulación de la expresión génica permite a las células adaptarse a los cambios en el ambiente.
La transcripción es el primer proceso de la expresión génica, mediante el cual se transfiere información del ADN hacia proteínas, usando ARN como mediador. La cadena de doble hélice que va a servir de codificadora depende de cada gen. Solo el 10 al 12% del ADN son genes.
Existen genes de tipo constitutivo y adaptativo.
Genes constitutivos: son genes que se expresan continuamente pues codifican para enzimas o proteínas necesarias para el metabolismo básico.
Genes adaptativos: son genes que se expresan solo en determinadas situaciones, por ejemplo cuando la célula se adapta a una determinada situación ambiental, y codifican para enzimas o proteínas que se necesitan en momentos concretos.
El código genético es universal (excepto en algunas bacterias), organizado en codones, específico (cada codón codifica un aminoácido), no es solapado, se lee en sentido 5’ 3’, redundante y no ambiguo (hay varios codones para un aminoácido)
ARN polimerasa
La ARN polimerasa es una enzima compuesta de múltiples cadenas polipeptídicas que cataliza la polimerización de NTP dirigida por un molde de ADN, siempre en dirección 5’-3’. Sin embargo, la ARN polimerasa no requiere de un cebador pues la transcripción comienza de novo en secuencias especificas al principio de los genes.
Aunque la ARN polimerasa es diferente en eucariotas y en procariotas, todas emplean mecanismos fundamentalmente similares para transcribir el ADN.
Transcripción en procariotas
La ARN polimerasa intacta se compone de cuatro subunidades denominadas: α, β, β’, ω y σ. La subunidad σ está unida de forma relativamente débil y puede separarse de las otras subunidades para buscar más promotores, o “quedarse pegada” a un promotor si es un gen que debe transcribirse constantemente.
Subunidad Funciónσ Reconocer al promotorΩ Ensamblaje de la enzima y regulación de la
expresión
β y β’Forman una pinza que sujeta el molde de ADN mediante enlaces fosfodiester (β’). Acomodan aproximadamente 20 pares de bases en su interior y contienen el centro activo (β).
α (x2) Ensamblaje de la enzima y regulación de la frecuencia de iniciación pues se unen a proteínas y secuencias reguladoras

Laura Mendoza~ 2 ~
Promotor
Es la secuencia de ADN a la que se une la ARN polimerasa para iniciar la transcripción de un gen.
Corriente arriba existen dos tramos de secuencias que son similares en muchos genes. Estas regiones abarcan seis nucleótidos cada una y se encuentran de -10 a -35 bases corriente arriba del sitio +1 (sitio de inicio).
Éstas secuencias no son idénticas entre distintos promotores, pero tienen una similitud suficiente para establecer secuencias de consenso, y su importancia funcional viene dada en que los genes con promotores que difieren de la secuencia consenso son transcritos de una forma menos eficiente y en que cualquier mutación introducida en cualquiera de las dos secuencias puede afectar la función promotora.
La ARN polimerasa por lo general se une a los promotores en una región de unos 60 pares de bases, que van desde el nucleótido -40 corriente arriba al nucleótido +20 corriente abajo. La subunidad σ se une a los sitios -35 y -10 por el tamaño de sigma.
Inicialmente el conjunto entre la ARN polimerasa y el promotor se denomina complejo promotor cerrado pues el ADN no está desenrollado. La polimerasa después desenrolla unas 12-14 bases de ADN alrededor de sitio de inicio, formando un complejo promotor abierto donde ya hay una hebra de ADN para servir como molde para iniciar la transcripción, agregándosele dos NTP. Tras la adición de unos 10 nucleótidos, σ se separa y la polimerasa progresa sobre el molde de ADN para continuar con la elongación.
La polimerasa desenrolla el ADN que tiene por delante y va enrollando el que queda atrás, manteniendo una región desenrollada de aproximadamente 15 pares de bases. En el interior de esta porción, 8-9 bases de la cadena creciente de ARN se encuentran unidas a la hebra de ADN molde.
Terminación de la transcripción
Cuando la ARN polimerasa encuentra una señal de terminación, se detiene la transcripción separándose el ARN, la polimerasa y el ADN.
Existen dos mecanismos para terminar la transcripción, el más simple y común consiste en una secuencia palindrómica rica en GC seguida de cuatro o más residuos de A. La secuencia palindrómica forma una horquilla y la transcripción llega a su fin. Se cree que la presencia de residuos de A facilita la disociación del ARN del molde pues los enlaces A-U son más débiles. Sin embargo, en algunos genes puede terminarse la transcripción gracias a una proteína llamada ρ, que se une a los segmentos mayores de 60 nucleótidos de ARN.
Control de la transcripción
La mayoría de la regulación transcripcional en bacterias ocurre en la iniciación, aunque también puede regularse en la elongación.
Los genes en procariotas están organizados en operones, los cuales son un grupo de genes estructurales cuya expresión está regulada por elementos de control o genes. Un operón consiste en:
Un operador: Controla el acceso de la ARN polimerasa al promotor.

Laura Mendoza~ 3 ~
Un gen regulador: Controla el tiempo y la velocidad de transcripción de otros genes. Éste gen codifica para una proteína que se une al operador, obstruyendo el promotor. Cuando se remueve la proteína represora, puede producirse la transcripción.
Un promotor. Un gen estructural: segmento de ADN que codifica para un polipéptido específico.
Los modelos de regulación de la transcripción pueden ser:
Inducibles: Donde la proteína reguladora es un represor que impide la expresión de genes estructurales en ausencia del inductor. (ejemplo: cuando no hay lactosa)
Reprimibles: Cuando un producto metabólico (ejemplo: triptófano) está en cantidades suficientes, se pueden dejar de fabricar las enzimas que lo sintetizan. En este sistema, el producto es el represor.
A su vez, el control de la transcripción puede ser negativo o positivo.
Control negativo de la transcripción: es cuando la proteína represora detiene la transcripción. Por ejemplo, el operón lac. Cuando no hay lactosa en el medio, el represor se une al operador y bloquea la transcripción de los genes estructurales.
Control positivo de la transcripción: es cuando el producto del gen regulador activa la expresión de los genes. Por ejemplo, la glucosa se usa de forma preferencial como fuente de energía, por lo tanto, si está disponible, no se expresan las enzimas implicadas en fuentes alternativas de energía. La glucosa reprime al operón lac incluso en presencia del inductor normal (lactosa). Esta represión positiva depende de los niveles de AMPc.
Transcripción en eucariotas
La transcripción en eucariotas es mucho más compleja que en procariotas. Esto se refleja en tres diferencias fundamentales:
Las células eucariotas contienen varias ARN polimerasas que transcriben distintas clases de genes.
Las ARN polimerasas en eucariotas interaccionan con un conjunto de proteínas específicas (factores de transcripción) para iniciar la transcripción.
La transcripción en eucariotas tiene lugar sobre la cromatina y no sobre el ADN libre.
Los factores de transcripción son esenciales para la transcripción pero no pueden aumentar o disminuir su ritmo.
ARN polimerasa en eucariotas
Las tres ARN polimerasas nucleares son enzimas complejas que se componen de 12 a 17 subunidades cada una.
ARN polimerasa Nro de subunidades Tipos de ARN sintetizados
I 13 subunidades -ARNr 28S, 18S, 5,8S
II 12 subunidades -Genes codificadores de proteínas (ARNm)

Laura Mendoza~ 4 ~
-Transcribir moléculas de ARNmi
III 17 subunidades -ARNr 5S
-ARNt (ARNtransferencia)
II y III -ARNsn y ARNsc
Mitocondriales -Genes mitocondriales
Cloroplásticas -Genes cloroplásticos
ARN polimerasa II
La polimerasa II solo es capaz de iniciar la transcripción si se añaden proteínas adicionales a la reacción, llamadas factores de transcripción. Aproximadamente el 10% de los genes codifica factores de transcripción.
Promotores
Los promotores de los genes transcritos por la polimerasa II contienen varias secuencias diferentes. El primer elemento descrito fue la caja TATA (TATAA) situada a 25-30 nucleótidos corriente arriba, sin embargo esta secuencia solo aparece en el 10-20% de los promotores de la ARN polimerasa II. Otras secuencias son el elemento iniciador (Inr), los elementos de reconocimiento por TFIIB (BRE) y varios elementos localizados corriente abajo (DPE, DCE, MTE).
Maquinaria de transcripción
Se requieren cinco (o seis) factores generales de transcripción para poner en marcha la transcripción por la polimerasa II (TFIIB, TFIID, TFIIE, TFIIF, TFIIH) y un complejo proteico mediador que consiste en más de 20 subunidades distintas que interaccionan con los factores generales de transcripción y con la ARN polimerasa.
Nombre Función Subunidades(TFIIA) Forma un complejo con TBP y la caja
TATA.TFIIB Actúa como puente para que la
polimerasa se una a TBP.Se une a la secuencia BRE.
TFIID TBP se une a la caja TATA y las otras subunidades se unen a Inr, DPE, DPC y MTE.
Proteína de unión a TATA (TBP)14 polipéptidos llamados factores asociados a TBP (TAF)
TFIIF Estabiliza la ARN polimerasa II mientras está en contacto con TBP y TFIIB
TFIIE

Laura Mendoza~ 5 ~
TFIIH Induce la liberación de la polimerasa mediante la fosforilación del CTD
Dos subunidades helicasas necesarias también para la escisión y reparación de nucleótidos.Una proteína quinasa que fosforila
Mediador Estimula la transcripción basal y desempeña una función en la asociación de los factores generales de transcripción con los factores específicos de transcripción de ciertos genes que regulan la expresión génica.
20 subunidades distintas
1. Se forma un complejo entre TFIID y el promotor. 2. Se une TFIIB a la subunidad TBP de TFIID y a la secuencia BRE para actuar
como puente entre la polimerasa y TBP.3. La polimerasa se une a TBP-TFIIB junto al tercer factor, TFIIF, que estabiliza el
complejo.4. Se unen TFIIE y TFIIH, completando el complejo de preiniciación.5. Una subunidad de TFIIH fosforila determinadas secuencias repetidas en el
dominio C-terminal de la polimerasa II (CTD), el cual consiste en repeticiones en tándem (27 en levaduras, 53 en humanos) de siete aminoácidos con la secuencia Tyr-Ser-Pro-Thr-Ser-Pro-Ser.
6. Las proteínas mediadoras se liberan de la polimerasa.7. La fosforilación del residuo 5 de serina induce la liberación de la polimerasa del
complejo de preiniciación y da lugar al comienzo de la transcripción.8. El CDT fosforilado se une a otros factores de elongación y procesamiento.
ARN polimerasa I
Ésta polimerasa transcribe genes de ARN ribosómico, que está presente en forma de repeticiones en tándem. La transcripción de estos genes da lugar a un pre-ARNr 45S de gran tamaño, que es procesado para dar los ARNr 28S, 18S y 5,8S. El promotor de los genes de ARN ribosómico se extiende unos 150 pares de bases corriente arriba del sitio de inicio de la transcripción. Estas secuencias son reconocidas por los factores de transcripción UBF y SL1, que se unen al promotor y reclutan a la polimerasa I.
El factor de transcripción SL1 contiene una subunidad TBP, aunque el promotor de los genes de ARN ribosómico no contiene una secuencia TATA.
ARN polimerasa III
Ésta polimerasa transcribe los genes del ARNt, ARNr 5S y MicroARN a partir de tres clases de promotores diferentes, dos de los cuales se encuentran en el interior de la secuencia transcrita.
ARNr 5S: 1. El factor TFIIIA se une al promotor.2. Se unen secuencialmente TFIIIC y TFIIIB y la polimerasa.
ARNt: Los promotores de ARNt no contienen la secuencia de ADN reconocida por el TFIIIA.
1. El factor TFIIIC se une a los promotores de los genes de ARNt.

Laura Mendoza~ 6 ~
2. Se unen secuencialmente TFIIIB y la polimerasa. MicroARN: Los promotores se localizan por delante del punto de iniciación de la
transcripción. Contienen una secuencia TATA y un punto de unión para otro factor llamado SNAP.
1. SNAP y TFIIIB se unen a los promotores (TFIIIB a la caja TATA mediante TBP)
2. TFIIIB recluta a la polimerasa.
Regulación de la transcripción
En eucariotas la transcripción también está controlada por proteínas que se unen a secuencias reguladoras específicas y modulan la actividad de la ARN polimerasa. Además, existen ARN no codificantes quienes también regulan la transcripción en las células mediante modificaciones de la estructura cromatínica.
Todas las células tienen el mismo genoma, pero existen más de 200 tipos de células, es decir que la diferenciación celular está basada en la regulación de la expresión de los genes. No todas las células fabrican las mismas proteínas.
Control transcripcional:
Secuencias reguladoras: Enhancers o intensificadoras : Secuencias reguladoras de acción en cis que
pueden estar localizadas a miles de nucleótidos corriente arriba o corriente abajo del promotor, en un cromosoma homólogo e incluso en un intrón. Se le unen proteínas activadoras (factores específicos de la traducción), quienes ayudan a determinar cuáles genes serán “encendidos” y la tasa de transcripción que tendrán. Existen aisladores que dividen cromosomas en dominios individuales de cromatina que no puede extenderse más allá del aislante y previenen que un enhancer en un dominio actúe sobre el promotor del dominio siguiente.
Las proteínas activadoras constan de dos dominios: una región que se une al ADN, la cual ancla el factor de transcripción en el sitio correcto del ADN; y otra región que activa la transcripción interactuando con otras proteínas, incluyendo el mediador u otros componentes de la maquinaria de transcripción.
Estos dominios de unión al ADN pueden ser:
Dominio de dedo de Zinc: contiene repeticiones cisteína e histidina que ligan iones de cinc y se pliegan en bucles que se unen al ADN. Los receptores de hormonas esteroideas, que regulan la transcripción en respuesta a hormonas como estrógeno y testosterona, poseen dominios en dedos de zinc.
Hélice-giro-hélice: Una hélice está en contacto con el ADN y las otras estabilizan la interacción.
Cremallera de leucina: contiene cuatro o cinco residuos de leucina separados por intervalos de siete aminoácidos. Inmediatamente después de esta cremallera existe una región rica en lisina y arginina, que se une al ADN.
Hélice-bucle-hélice: es similar a la cremallera de leucina, pero sus dominios de dimerización están formados por dos regiones helicoidales, separados por un bucle.

Laura Mendoza~ 7 ~
Distintos miembros de cremalleras de leucina y hélices-bucles-hélices pueden formar dímeros, teniendo una importante función en la especificidad tisular.
Los dominios activadores de estos factores de transcripción pueden ser ricos en Aspartato y glutamato, otros pueden ser ricos en prolina o glutamina.
Los factores de activación interactúan con varios coactivadores que estimulan la transcripción modificando la estructura de la cromatina
Silencers o silenciadoras: Secuencias reguladoras de acción en cis que pueden estar localizada a miles de nucleótidos del promotor, que hacen más lenta la transcripción. Se les unen proteínas represoras (factores específicos de la traducción)
Algunas proteínas represoras pueden simplemente interferir con la unión de otros factores transcripcionales al ADN, otras compiten con los activadores por unirse a secuencias específicas de regulación. Otros contienen dominios específicos que inhiben la transcripción por medio de interacciones proteína-proteína (proteínas activadoras específicas, con el mediador, con factores de transcripción generales o con co-represores que modifican la estructura de la cromatina)
Los dominios represores pueden ser ricos en residuos de alanina, prolina o residuos acídicos.
Regulación de la elongación: Tras el comienzo de la transcripción, la polimerasa sintetiza una secuencia corta de ARN y se detiene en un sitio cercano al comienzo del gen, aproximadamente a unos 50 nucleótidos del sitio de inicio. Esta parada obedece a la asociación de factores negativos como NELF y DSIF. La reanudación del proceso depende de la acción de otro factor, llamado P-TEFb, el cual fosforila a los factores NELF, DSIF y a la serina del CDT de la polimerasa.
Modificación de la cromatina:
Las proteínas HMG afectan la estructura de la fibra de la cromatina. Se conocen tres familias de estas proteínas: HMGA, HMGB y HMGN. Las proteínas HMGA y HMGB pueden curvar la molécula de ADN para facilitar la unión de diversos factores reguladores de la cromatina. HMGN se unen a los nucleosomas en sitios que se solapan con la unión de la H1 y pueden inducir a la descondensación de la estructura de la cromatina.
Las modificaciones de las histonas influyen en la expresión génica tanto como consecuencia de la alteración de las propiedades de la cromatina como al crear sitios de unión de otras proteínas que activan o inhiben la transcripción. Éstas modificaciones se regulan entre si, es decir, un cambio que favorezca la activación puede inducir otro cambio que favorezca la activación y éste inhibe un cambio que favorezca la represión. O por el contrario, un cambio que favorezca la represión puede inhibir un cambio que favorezca la activación. Esto es un mecanismo de herencia epigenética pues las histonas modificadas pasan de los cromosomas parentales a los cromosomas hijos.

Laura Mendoza~ 8 ~
Acetilación de histonas: favorece la transcripción. Las histonas están cargadas positivamente, al acetilarlas se neutralizan sus cargas positivas y reduce la capacidad de unirse al ADN, expandiendo la cromatina. Algunos represores genéticos desacetilan las histonas y algunos activadores genéticos las acetilan. También regulan la reparación del ADN. Las histonas acetiltransferasas se asocian a coactivadores transcripcionales y a TFIID. Los residuos acetilados de lisina actúan como sitio de unión de muchos otros factores activadores.
Metilación de histonas: Puede estar relacionado con la activación o la represión de la transcripción. Algunas proteínas activadoras se unen a residuos metilados de lisina-4, mientras que la metilación de las lisinas 9 y 27 de H3 se asocian a la condensación y represión de la cromatina.
Fosforilación de histonas: por lo general está relacionado con la activación transcripcional.
La elongación transcripcional a través de la cromatina se ve facilitada por los factores de elongación asociados al dominio C-Terminal fosforilado de la polimerasa II. Entre estos factores figuran enzimas modificadoras de histonas (acetiltransferasas y metiltransferasas) y factores remodeladores de la cromatina, quienes son complejos proteicos que aprovechan la energía de la hidrolisis del ATP para alterar los contactos del ADN con las histonas. Los mecanismos de acción de estos factores pueden ser:
o El desplazamiento de las histonas a lo largo de la molécula de ADN. o La inducción de cambios conformacionales en los nucleosomas: afectando la
capacidad de secuencias específicas de ADN de interaccionar con proteínas reguladoras transcripcionales.
o Eliminar histonas del ADN.
Metilación del ADN: Los residuos de citosina que preceden a guaninas en el ADN son modificados por la adición de grupos metilos en el carbono 5 para reprimir la transcripción. Es una forma de herencia epigenética.
Un papel regulador importante de la metilación del ADN es la impresión genómica o imprinting, que controla la expresión de algunos genes del desarrollo embrionario, pues existen algunos genes cuya expresión depende de si se heredan de la madre o del padre.
Regulación por ARN no codificante: Moléculas de ARNsi (de interferencia) y MicroARN suelen inhibir la traducción o inducir la degradación de ARNm homólogos a través de la inducción de modificaciones de histonas. Los ARNsi se asocian a un complejo proteico RITS (complejo de silenciamiento transcripcional inducido por ARN) que contiene proteínas que inducen la condensación cromatínica y la metilación de la lisina-9 de H3. También puede inducir la degradación de un ARNm mediante el complejo RISC.
Aminoácidos fosforilables:
treonina, serina, tirosina
Aminoácidos acetilables: lisina
Aminoácidos metilables: lisina y
arginina

Laura Mendoza~ 9 ~
Inactivación del cromosoma X
El cromosoma X contiene unos 1000 genes que no están presentes en el cromosoma Y, sin embargo, las células femeninas y masculinas contienen cantidades equivalentes de las proteínas codificadas por genes del cromosoma X, pues existe un mecanismo de compensación de la dosis, en el que la mayoría de los genes de uno de los cromosomas X están inactivados tras ser convertidos en heterocromatina en un estadio temprano del desarrollo.
Éste mecanismo tiene su clave en un ARN no codificante transcrito a partir de un gen regulador llamado Xist, el cual recluta un complejo proteico que induce la metilación de la lisina-27 y 9 de la H3, provocando la condensación de la cromatina.
Control post-transcripcional: Empalme alternativo: Dos moléculas de ARNm son procesadas de manera
diferente a partir del mismo gen.

Laura Mendoza~ 10 ~
Maduración y renovación del ARN
La mayor parte del ARN de nueva síntesis debe ser modificado para convertirse en su forma funcional, exceptuando el ARNm bacterianos, pues se usa mientras están siendo transcritos. La maduración de ARNr y ARNt es similar en eucariotas y procariotas.
ARN ribosómico
Las procariotas poseen tres ARNr (23S, 16S, 5S), los cuales son obtenidos a partir de un pre-ARN único.
Las eucariotas poseen cuatro tipos de ARNr (28S, 18S, 5,8S y 5S), el único que no es casi procesado es el ARNr 5S pues se transcribe a partir de un gen distinto, mientras que los otros tres provienen de un pre-ARN común.
Maduración del ARNr en bacterias:
1. Corte inicial del pre-ARN produce precursores separados para los tres ARNr.2. Se producen escisiones secundarias hasta obtener los productos finales.
Maduración en eucariotas: tiene lugar en el nucléolo.
1. Corte inicial en un sitio adyacente al ARNr 5,8S en su extremo 5’, dando dos precursores que contienen el ARNr 18S y el ARNr 28S y 5,8S
2. Escisiones posteriores dan lugar a los productos finales.3. El ARNr 5,8S se une por puentes de H a la molécula de 28S
ARN de transferencia
Los ARNt eucarióticos y bacterianos son sintetizados como pre-ARNt, algunas de las cuales contienen secuencias de varios ARNt individuales.
En bacterias, algunos ARNt proceden de los transcritos pre-ARNr.
El procesamiento del extremo 5’ de los pre-ARNt se realiza gracias a una ribozima llamada ARNasa P, la cual contiene ARN como centro catalítico y moléculas proteínicas sin las cuales no tiene el nivel de actividad máximo.
El procesamiento del extremo 3’ de los ARNt se forma por la acción de una ARNasa proteica, que, en caso de que no lo tenga ya, añade una secuencia terminal CCA.
Aproximadamente un 10% de las bases de ARNt son alteradas para dar una serie de nucleótidos modificados en posiciones específicas. Algunos pre-ARNt tienen intrones que son eliminados por corte y empalme mediado por enzimas proteicas convencionales
ARN mensajero en eucariotas
Las principales diferencias entre la maduración del ARNm en procariotas y eucariotas son que en las bacterias la traducción ocurre de forma inmediata; que en eucariotas el ARNm

Laura Mendoza~ 11 ~
debe ser transportado al citoplasma y que el pre-ARNm en eucariotas debe ser ampliamente modificado en ambos extremos y se deben eliminar los intrones.
Estas reacciones están acopladas a la transcripción y el dominio CTD de la pol II sirve como sitio de unión para los complejos enzimáticos implicados en la maduración. Ésta asociación es responsable de la especificidad de la pol II en el procesamiento del ARNm. Las pol I y III no tienen CTD, por lo que sus productos no son procesados por los mismos complejos enzimáticos.
Pasos:
1. Modificación del extremo 5’ mediante la adición de una caperuza o cap de 7-metilguanosina: Ésta caperuza se forma por la adición de una molécula de GTP inversa al nucleótido 5’ terminal, luego se añaden grupos metilo a este residuo de G y a las ribosa de uno o dos nucleótidos 5’. Las enzimas responsables de esta reacción son reclutadas al CTD fosforilado y la caperuza es añadida tras la transcripción de 20-30 nucleótidos. Ésta caperuza estabiliza el ARN y alinea los ARNm eucarióticos sobre el ribosoma.
2. El extremo 3’ de la mayoría de los ARNm eucarióticos se forma por el corte del transcrito primario y la adición de una cola de poli-A (poliadenilación). Las señales para este proceso incluyen un hexanucleótido (AAUAAA) localizado entre 10 y 30 nucleótidos corriente arriba del sitio de poliadenilación y un elemento de secuencias ricas en G-U corriente abajo, aunque algunos genes tienen estas secuencias de G-U corriente arriba de AAUAAA. Estas secuencias son reconocidas por un grupo de proteínas, incluyendo una endonucleasa que corta la cadena de ARN y una poli-A polimerasa que añade una cola de unos 200 nucleótidos. Éstas enzimas se asocian con el CTD fosforilado y pueden viajar con la polimerasa desde la iniciación. Las colas de poli-A regulan la traducción y la estabilidad del ARNm.
Ambas modificaciones protegen al ARN de la degradación.
Corte y empalme (splicing)
La mayor parte de los genes están interrumpidos por múltiples secuencias no codificantes llamadas intrones que son escindidas de forma precisa del ARN maduro. Los pre-ARNm contienen múltiples exones cortos (150 nucleótidos en humanos) separados por intrones de aproximadamente 3500 nucleótidos.
Las señales para el corte y empalme son secuencias consenso en cada sitio de corte y de ramificación.
El splicing tiene lugar en complejos llamados espliceosomas, compuestos de proteína y ARN. Estos ARN, llamados ARN pequeños nucleares (ARNsn) son de cinco tipos: U1, U2, U4, U5 y U6, que tienen un tamaño de entre 50 y 200 nucleótidos, formando un complejo con entre seis y diez moléculas proteínicas para dar RNPsn.

Laura Mendoza~ 12 ~
Las RNPsn U1, U2 y U5 contienen cada una una molécula única de ARNsn, mientras que las RNPsn U4 y U6 están unidas entre sí formando una única RNPsn.
1. RNPsn U1 se une al sitio de corte 5’ (secuencia GU). 2. RNPsn U2 se une al punto de ramificación y a U1, convirtiendo al ARN en una
especie de búmeran.3. Se incorpora U4/U6 y U5. La U5 se une a secuencias corriente arriba del punto
de corte 5’ y a U1. U4/U6 se une a sitio de corte 3’ (secuencia AG).4. El pre-ARNm es cortado en el sitio de corte y empalme 5’.5. U4 y U1 se disocian del complejo, y U6 sustituye a U1 y hace un complejo con
U2, poniendo en contacto el punto de ramificación (un nucleótido de adenina cerca del extremo 3’) y el extremo 5’ del intrón, formándose un bucle por el vínculo inusual entre el extremo 5’ del intrón y el grupo hidroxilo 2’ de la adenina.
6. U5, aún unido a las secuencias corriente arriba del punto de corte 5’, se une a secuencias del sitio de corte 3’, manteniéndose la alineación entre los exones 5’ y 3’
7. Se corta en 3’ y se unen los dos exones. El intron es escindido como un lazo y es degradado en el núcleo.
El reconocimiento de los sitios de corte se realiza por apareamiento de bases entre la secuencia del consenso del 5’ y una secuencia complementaria en el ARNsn. Sin embargo, los intrones contienen muchas secuencias parecidas a los puntos de corte y empalme, la identificación de los lugares correctos de splicing se logra mediante proteínas llamadas factores de corte y empalme, que se unen a secuencias específicas de ARN en exones y reclutan las RNPsn U1 y U2 mediante interacciones proteína-proteína.
Los factores de corte y empalme también acoplan el corte y empalme a la transcripción mediante su asociación con el CTD fosforilado de la pol II, para asegurar que los exones se unan en el orden correcto a medida que se sintetiza el pre-ARNm.
Los ARNsn pueden catalizar la reacción de corte y empalme de forma directa. Es decir, determinados intrones en mitocondrias, cloroplastos y bacterias pueden autoempalmarse. U2 y U6 pueden catalizar ambos pasos del corte y empalme del pre-ARNm en ausencia de proteínas.
Corte y empalme alternativo
Se puede regular la expresión génica por medio del control de la maquinaria celular que lleva a cabo el corte y empalme. Se pueden producir distintos ARNm partiendo del mismo gen combinando los sitios de corte. Por ejemplo, más del 50% de los genes humanos producen transcritos que sufren corte y empalme alternativo, que podría aumentar la diversidad de proteínas que pueden ser codificadas pues los patrones de corte y empalme alternativos pueden variar entre los distintos tejidos y en respuesta a señales extracelulares.

Laura Mendoza~ 13 ~
Un gran grupo de proteínas regulan el corte y empalme mediante la unión a secuencias silenciadoras o activadoras.
Corrección del ARN
Son los procesos de maduración que alteran las secuencias codificadoras de proteínas de algunos ARNm. La corrección del ARNm implica cambios de bases únicas, como la desaminación de citosina a uridina y de adenosina a inosina.
Un ejemplo de desaminación de citosina a uridina es la corrección que da lugar a dos formas distintas de la apolipoproteína B. Una forma de esta proteína es más corta, por lo tanto tienen funciones diferentes, una se encuentra en el hígado y la otra en el intestino.
Un ejemplo de desaminación de la adenosina en inosina tiene como resultado cambios sencillos de aminoácidos en los receptores de algunas moléculas señalizadoras en las neuronas, generando potencialmente 24 versiones diferentes del receptor. La importancia de esto en el sistema nervioso radica en que la carencia de la enzima editora provoca defectos neurológicos en ratones y moscas.
Degradación del ADN
La mayoría de las secuencias transcritas en pre-ARNm son degradadas en el interior del núcleo, pues más del 90% de estas secuencias son intrones. Estos intrones se degradan gracias a una enzima que reconoce el enlace característico del punto de ramificación y gracias a otras enzimas que reconocen extremos 5’ o 3’ de ARN, catalizando la degradación en cualquier dirección.
Las células poseen un mecanismo de control de calidad que detecta y degradan ARNm aberrantes generados por error. Uno de estos mecanismos de control es el decaimiento de ARNm mediado sin sentido, degradando ARN que carecen de marcos completos de lectura abierta, evitando la síntesis de proteínas anómalas. Éste método se pone en marcha cuando los ribosomas detectan codones de terminación prematuros, interrumpiendo la traducción y degradando la molécula defectuosa de ARNm.
La degradación citoplasmática de la mayoría de los ARNm eucarióticos comienza con el acortamiento de las colas poli-A, luego se degradan por nucleasas que actúan a partir del extremo 3’ o la eliminación de la caperuza en el extremo 5’.
Los ARN degradados con rapidez contienen secuencias específicas ricas en AU cerca del extremo 3’ que actúan como sitios de unión a proteínas que pueden estabilizar o marcar los ARNm para inducir su degradación. Las actividades de estas proteínas se regulan por medio de señales extracelulares como factores de crecimiento u hormonas, haciendo a las células capaces de controlar la velocidad de degradación de ciertos ARNm como respuesta a las condiciones ambientales.
Los ARNsi y ARNmi controlan la estabilidad, degradación y traducción del ARNm

Laura Mendoza~ 14 ~
El nivel intracelular de ARN está determinado por un equilibrio entre la síntesis y la degradación. Tanto los ARNr como los de transferencia son muy estables, por lo tanto hay niveles elevados de ambos ARN en la célula, de aproximadamente 90%.
Los ARN bacterianos se degradan en tan solo 2 o 3 minutos, para que la célula pueda responder rápidamente a modificaciones de su entorno. En células eucariotas, el ARNm tiene una vida de entre 30 minutos hasta 20 horas.
Los ARNm inestables suelen codificar para proteínas reguladoras, como factores de transcripción, mientras que los ARNm que codifican para proteínas estructurales tienen vidas más largas.

Laura Mendoza~ 15 ~
Traducción del ARNm
La traducción es la síntesis de proteínas guiada por un molde de ARNm. Todos los ARNm se leen en dirección 5’-3’ y las cadenas polipeptídicas se sintetizan desde el extremo amino terminal al carboxilo terminal, donde cada aminoácido es codificado por un codón.
ARN de transferencia
Las distintas moléculas de ARNt sirven como adaptadores en el proceso de alineación de aminoácidos con el ARNm molde. Tienen una longitud de aproximadamente 70-80 nucleótidos con una estructura en forma de hoja de trébol debido a apareamiento de bases intracatenarios. Puede tener timina e incluso una base poco común: la inosina. Su plegamiento en forma de L es necesario para el correcto anclaje de los ribosomas.
Para poder cumplir su función, el ARNt necesita de dos regiones distintas y separadas, además de otros dos brazos
Brazo aceptor: la región de unión al aminoácido está formada por una secuencia CCA ubicada en el extremo 3’ donde los aminoácidos se unen covalentemente al –
OH de la adenosina. Bucle búmeran: es el lazo de reconocimiento del ARNm, es una región que
reconoce al ARNm mediante complementariedad de bases por un lazo anticodón. Brazo TψC: es el reconocimiento del ribosoma. Brazo D: su secuencia es reconocida por una de las 20 enzimas (aminoacil ARNt
sintetasas) muy específicas que unen cada aminoácido con su molécula de ARNt.
La incorporación del aminoácido correcto depende de la unión de cada uno al ARNt, asi como de la especificidad del apareamiento entre codón y anticodón. La sintetasa reconoce secuencias de nucleótidos específicas (incluyendo el anticodón) que son únicas de cada ARNt. Si el aminocil AMP es incorrecto, es hidrolizado antes de unirse al ARNt.
1. El aminoácido es activado mediante una reacción con ATP, donde es convertido a AMP, formándose un intermediario aminocil AMP.
2. El aminocil AMP se une al extremo 3’ del ARNt.3. El aminoácido se alinea en el ARNm molde por la complementariedad de bases
entre codón y anticodón.
La complementariedad de bases codón-anticodón es mucho menos estricta que la complementariedad regular, pues el código genético es redundante. De los 64 codones posibles, 3 son codones de terminación y 61 codifican para aminoácidos, provocando que un aminoácido sea codificado por más de un codón. Además de que algunos ARNt son capaces de reconocer más de un codón en el ARNm, como resultado de un reconocimiento no estándar entre el anticodón del ARNt y la tercera base de algunos codones pues hay un apareamiento de bases débil. Si la inosina aparece en el anticodón, un ARNt puede reconocer 3 codones en el ARNm.

Laura Mendoza~ 16 ~
Anticodón ARNt 5’ Codón ARNm 3’A UU A o GC GG U o C I U, C o A
Ribosomas
Son partículas subcelulares donde se sintetizan las proteínas en células eucariotas y procariotas. Una célula de mamífero puede contener aproximadamente 10 millones de ribosomas. Los ribosomas se pueden ensamblar in vitro.
Los ribosomas, al igual que el ARNt, tienen apareamientos de bases intracatenarios.
Eucariotas (r80S) Procariotas (r70s)Subunidad Composición Subunidad Composición
40S (pequeña) - ARNr 18S- 30 proteínas
30S (pequeña) - ARNr 16 S- 21 proteínas
60S (grande) - ARNr 28S, 5,8S y 5S- 45 proteínas
50S (grande) - ARNr 23S y 5S- 34 proteínas
La subunidad grande de los ribosomas es una ribozima, pues es capaz de catalizar la formación de enlaces peptídicos. La falta de proteínas ribosómicas provoca un descenso pero no una pérdida completa de la actividad ribosómica, por lo que se deduce que las proteínas ribosomales tienen una función estructural.
Señal de inicio de la traducción
La traducción no empieza simplemente en el extremo 5’ del ARNm ni termina simplemente en el extremo 3’, pues ambos bordes del ARNm son secuencias no codificantes (llamadas regiones UTR). Las señales que identifican el codón de iniciación son distintas en células eucariotas y procariotas, debido a las diferencias existentes entre los ARNm policistrónicos y monocistrónicos.
La traducción siempre comienza con metionina, codificado generalmente por el codón AUG. En algunas bacterias existen codones de iniciación alternativos, como el codón GUG que cuando se ubica al inicio de una cadena polipeptídica se incorpora metionina en lugar del aminoácido que codifica normalmente (valina)
En la mayoría de las bacterias, la síntesis se inicia con un residuo modificado de metionina (N-formilmetionina), a diferencia de las células eucariotas donde la metionina se encuentra sin modificar a excepción de en mitocondrias y cloroplastos.
Los codones de iniciación en los ARNm bacterianos son precedidos por la secuencia Shine-Dalgarno, la cual es una secuencia específica complementaria de una secuencia del ARNr 16S cerca del extremo 3’, que aproxima el ARNm al ribosoma.

Laura Mendoza~ 17 ~
En eucariotas, por el contrario, los ribosomas se unen al ARNm reconociendo la 5’-cap y se desplazan por la secuencia del ARNm hasta encontrar un codón de iniciación AUG. Las secuencias que flanquean al codón AUG también influyen en la iniciación, pues a veces el primer AUG en el ARNm no es reconocido y la traducción comienza en un triplete más alejado del extremo 5’.
Varios ARNm poseen sitios de entrada internos para ribosomas en los que puede iniciarse la traducción mediante la unión de un ribosoma a una posición interna del ARNm. Éste es un mecanismo de inicio que se activa en condiciones de estrés celular que permiten la traducción selectiva cuando se encuentra bloqueado el modo normal de inicio a partir de la 5’-cap.
Mecanismo de traducción
La traducción generalmente tiene tres etapas: iniciación, elongación y terminación. En general, la traducción comienza con la unión de un metionil ARNt específico y el ARNm a la subunidad menor del ribosoma. Luego, la subunidad mayor se une al complejo, formando un ribosoma funcional sobre el que se elonga la cadena polipeptídica. También son necesarias proteínas no ribosomas específicas.
Los ARNm son traducidos por una serie de ribosomas separados por aproximadamente 100 a 200 nucleótidos. Este conjunto de ribosomas se llama polisoma, donde cada ribosoma funciona de manera independiente.
Inicio
En procariotas
1. La subunidad 30S se une a dos factores de iniciación, IF1 e IF3 2. El factor de iniciación IF2-GTP, el ARNm y el N-formilmetionil ARNt se unen a este
complejo. Donde IF2 interacciona con el ARNt iniciador.3. El ARNt se une al codón de iniciación del ARNm y se liberan IF1 e IF3.4. La subunidad 50S se une al complejo, induciendo la hidrólisis del GTP y liberando
el GDP junto a IF25. Se forma un complejo 70S-ARNm-ARNt preparado para catalizar la formación de
un enlace peptídico en la fase de elongación.
En eucariotas requiere de al menos 11 proteínas distintas de múltiples cadenas polipeptídicas llamadas eIFs, estos factores reconocen tanto al extremo 3’ como al extremo 5’.
1. eIF1, eIF1A, eIF3 y eIF5 se unen a la subunidad 40S y el eIF2-GTP se une al metionil ARNt iniciador.
2. Se asocia la subunidad 40S y el ARNt iniciador (junto a todos los factores de iniciación).
3. La 5’-cap del ARNm es reconocido por eIF-4E, que forma un complejo con eIF-4A y eIF-4G, quien se une a la PABP asociada a la cola poli-A del extremo 3’.

Laura Mendoza~ 18 ~
4. Los factores de iniciación eIF-4E y eIF-4G en asociación con eIF-4A y eIF4B dirigen al ARNm hacia la unidad ribosómica 40S mediante la interacción entre los factores eIF-4G y el eIF-3 (se libera PABP).
5. La subunidad 40S unida al metionil ARNt y a los eIF chequea el ARNm hasta identificar un codón AUG, este desplazamiento requiere energía.
6. El codón se reconoce y eIF5 provoca la hidrólisis del GTP unido a eIF2, liberando todos los factores de iniciación y permitiendo que eIF5B facilite la unión de la subunidad 60S usando GTP para iniciar el complejo de iniciación 80S.
Elongación: El ribosoma tiene tres sitios de unión del ARNt, llamados P (peptidil), A (aminoacil) y E (liberación).
1. El metionil ARNt iniciador se une al sitio P. 2. Un factor de elongación (EF-Tu en procariotas y eEF1α en eucariotas) unido a
GTP guía al aminoacil ARNt hacia el ribosoma, donde se une al sitio A mediante emparejamiento con el segundo codón del ARNm.
La subunidad pequeña tiene un centro descodificador que reconoce los pares de base codón-anticodón correcto.
3. La inserción del aminoacil ARNt en el dominio A provoca un cambio conformacional que induce la hidrólisis del GTP y la liberación de eEF1α-GDP.
4. Se forma un enlace peptídico entre el metionil ARNt iniciador del sitio P y el aminoacil ARNt del sitio A.
5. Se transfiere la metionina al aminoacil ARNt formándose un peptidil ARNt en el sitio A y dejando un ARNt libre en el sitio P.
6. EF-G en procariotas y eEF-2 en eucariotas unido a GTP se hidroliza e induce la translocación, desplazando el ribosoma tres nucleótidos sobre el ARNm, moviendo el peptidil ARNt al sitio P, colocándose un nuevo codón en el sitio A libre y ubicando el ARNt libre en el sitio E.
7. La unión de un nuevo aminoacil ARNt al sitio A induce la liberación del ARNt libre.8. eEF1βγ en eucariotas y EF-Ts en procariotas sustituye el GDP unido a eEF1α o
EF-Tu por GTP, para que el factor pueda dirigir un nuevo aminoacil ARNt al sitio A.9. La elongación continúa.
Terminación1. Un codón de terminación (UAA (ocre), UAG (ámbar) o UGA (ópalo)) se coloca en
el sitio A. Los ARNt no contienen anticodones complementarios de los tripletes de terminación, pero tienen factores de liberación (RF1 y RF-2 en procariotas y eRF1 en eucariotas) y RF3 en procariotas y eRF3 en eucariotas, que no reconocen codones de terminación pero se asocian a RF1-RF2 o eRF1.
2. El factor de liberación se une a un codón de terminación en el sitio A, hidrolizando el enlace entre el ARNt y la cadena del sitio P, liberándola.
3. El ARNt es liberado, y las subunidades del ribosoma y el ARNm se disocian.
Regulación de la traducción

Laura Mendoza~ 19 ~
La traducción es regulada en respuesta al estrés celular, la disponibilidad de nutrientes y la estimulación por proteínas represoras de la traducción y microARN no codificantes.
Unión de proteínas represoras, que bloquean a secuencias específicas del ARNm. Por ejemplo, en ausencia de hierro, proteínas reguladoras se unen a una secuencia de respuesta a hierro en el extremo 5’ no codificante, interfiriendo en la unión del ARNm a la subunidad 40S. En presencia de hierro, la proteína reguladora no se une, de modo que puede comenzar la traducción.
La traducción puede ser regulada mediante proteínas en secuencias de las regiones 3’ no codificantes. En algunos casos, los represores se unen al factor eIF4E e interfieren en la interacción de eIF4E y eIF4G. Estas proteínas que se unen al extremo 3’ también son responsables de la localización del ARNm en regiones específicas de la célula. Largo de la cola poli A: Muchos ARNm son almacenados sin traducir con
pequeñas colas de poli A, de aproximadamente 30-50 nucleótidos. En un principio, poseen colas largas de aproximadamente 200 nucleótidos, y luego son reconocidas por una proteína represora que se une a secuencias específicas de sus regiones no codifcantes del extremo 3’ y escinden la mayor parte de las colas poli-A. Si se necesitan, estos ARNm son incorporados a la traducción mediante el alargamiento de sus colas poli-A, permitiendo la unión de la PABP.
Interferencia de ARN (iARN): inhibe a través de la inducción de formación de heterocromatina o induciendo la degradación de moléculas diana de ARNm.
ARNsi: se tiene un ARN bicatenario que es escindido por una enzima llamada Dicer, formando ARNsi.
ARNmi: la polimerasa II transcribe pre-ARNmi de unos 70 nucleótidos, que son sometidos a la escisión secuencial mediante nucleasas Drosha y Dicer para la formación de una molécula de unos 22 nucleótidos. El genoma humano codifica para unos 1000 ARNmi y se cree que cada uno puede reconocer hasta 100 ARNm y que tienen un papel en el desarrollo embrionario temprano, (sistema nervioso, musculatura, corazón, pulmones y sistema inmunitario). Estos ARN regulan la proliferanción y supervivencia de la célula, la expresión anómala de estos ARN se asocia a cardiopatías y diversos tumores.
El ARN o ADN de cadena única antisentido son complementarios al ARNm de un gen de interés, formando hibridos con ARNm diana, bloqueando la traducción.
1. ARNsi o ARNmi se asocia con un complejo de proteínas llamado complejo silenciador inducido por ARN (RISC)2. Las dos hebras del ARNsi o ARNmi se separan formando una hebra antisentido, la cual dirige a RISC a un ARNm diana, inhibiendo la traducción.3. El ARNm es escindido por una de las proteínas de RISC.4. El complejo RISC-ARNsi/ARNmi se disocia. Modulación de la actividad de factores de iniciación:

Laura Mendoza~ 20 ~
El eIF-2 y el eIF2B pueden ser fosforilados por proteínas quinasas reguladoras, inhibiendo el intercambio de GDP por GTP.
Factores de crecimiento que estimulan la síntesis activan proteínas quinasas que fosforilan proteínas 4E-BPs, quienes se unen a eIF-4E. En ausencia de dichos factores, las 4E-BPs no fosforiladas se unen a eIF-4E e inhiben la traducción.
Plegamiento de proteínas
La síntesis del polipéptido no significa que la proteína sea funcional, pues ésta debe plegarse correctamente en su estructura terciaria para cumplir su función.
Chaperonas
Son proteínas que actúan como catalizadoras que facilitan el ensamblaje sin formar parte del complejo ensamblado y sin proporcionar ningún tipo de información adicional. En ausencia de las chaperonas las proteínas se pliegan incorrectamente o se agregan formando complejos insolubles relacionados a enfermedades como Alzheimer o Parkinson.
Las chaperonas se unen a las cadenas polipeptídicas nacientes mientras se sintetizan en los ribosomas, previniendo el plegamiento incorrecto antes de que la síntesis finalice. También estabilizan los polipéptidos no plegados durante su transporte a orgánulos. El número de chaperonas en células eucariotas es considerablemente mayor que en procariotas.
Hay chaperonas en el citosol y chaperonas en el interior de los orgánulos, por ejemplo, las chaperonas mitocondriales, quienes facilitan la transferencia del polipéptido al atravesar la membrana y su plegamiento posterior.
Existen dos familias de chaperonas, las Hsp (Heat-shock proteins) y las chaperoninas. Las Hsp70 estabilizan las proteínas durante la traducción y el transporte, uniéndose a siete u ocho residuos de aminoácidos, manteniendo el polipéptido en una conformación extendida. La cadena entonces es transferida de una chaperona Hsp70 a una chaperonina, en cuyo interior tiene lugar el plegamiento.
Las chaperoninas consisten en multiples subunidades proteicas organizadas en dos anillas apiladas para formar una estructura con dos cámaras (como un barril). Dentro, las cadenas no plegadas están resguardadas del citosol, permitiendo el plegamiento y evitando que se unan otros polipéptidos no plegados.
Enzimas
Catalizan el plegamiento mediante rotura y formación de nuevos enlaces covalentes.
La proteína disulfuro isomerasa (PDI): cataliza la formación de puentes disulfuro, los cuales son especialmente abundantes en proteínas secretadas y algunas proteínas de membrana, pues el citosol contiene agentes reductores que impiden

Laura Mendoza~ 21 ~
la formación de puentes disulfuro. En eucariotas, los puentes disulfuro se forman dentro del retículo endoplasmático, cuyo lumen es un ambiente oxidante. La PDI es una de las proteínas más abundantes del RE.
Peptidil prolil isomerasa: cataliza la isomerización de los enlaces peptídicos donde participa la prolina, quien puede adoptar una configuración cis o trans, formando curvaturas o codos en la cadena. La isomerasa requiere que la siguiente serina o treonina estén fosforiladas.
Maduración de proteínas
Escisión de proteínas (proteólisis): La proteólisis es una etapa importante en la maduración de muchas proteínas.
Eliminación de la metionina inicial de muchos polipéptidos, que ocurre antes de que la traducción de la cadena se sintetice completamente.
Escisión de la secuencia señal que marca a las proteínas que se dirigen al retículo endoplasmático, a los lisosomas o a la membrana y tiene una gran importancia en la translocación.
Escisión de precursores inactivos de mayor tamaño para formar enzimas u hormonas funcionalmente activas. Por ejemplo la insulina, que se forma a partir de dos escisiones de un precursor llamado preproinsulina; la formación de las enzimas digestivas, las proteínas que participan en la coagulación sanguínea, y de proteasas que regulan la muerte celular programada. También es importante en la replicación del VIH, donde una proteasa codificada por el virus escinde precursores para formar proteínas víricas estructurales.
Glicosilación: es la adición de carbohidratos. Las proteínas a las que se añaden una cadena de glúcidos (glicoproteínas) son secretadas o se localizan en la superficie celular, aunque algunas proteínas nucleares o citosólicas también están glicosiladas. Se inicia en el RE antes de que termine la traducción. Los carbohidratos de las glicoproteínas sirven para evitar la agregación de proteínas en el RE, en la decisión del destino de las proteínas y como reconocimiento en interacciones célula-célula.
N-glicoproteínas: los carbohidratos (N-acetilgluocosamina) se unen al N de la cadena de asparragina.
1. Un oligosacárido de 14 residuos de monosacáridos (dos N-actetilglucosamina, nueve manosas y tres glucosas) que estaba ubicado en la membrana del RE unido a un lípido transportador, el dolicol fosfato, se transfiere a un residuo de asparragina de la secuencia Asn-X-Ser o Asn-X-Thr (X puede ser cualquiera menos prolina) mediante una enzima llamada oligosacaril transferasa.
2. Tres residuos de glucosa son eliminados en el RE.3. Se eliminan y se añaden otros azúcares en el aparato de Golgi.
O-glicoproteínas: los carbohidratos (N-acetilgalactosamina) se unen al átomo de O de la serina o treonina.

Laura Mendoza~ 22 ~
1. En el aparato de Golgi se añaden los O-oligosacáridos formados por la adición de un único azúcar de pocos monosacáridos (pudiendo ser galactosa, ácido siálico o N-acetilgalactosamina)
Los residuos de manosa pueden unirse a residuos de triptófano a través de enlaces C-C. Anclaje de lípidos a cadenas polipeptídicas: estas modificaciones marcan y anclan
las proteínas a la membrana plasmática. o Cara citosólica: N-miristoilación: el ácido mirístico (14C) se une al residuo N-terminal de
glicina, el cual es frecuentemente el segundo aminoácido incorporado a la cadena.
Prenilación: grupos prenilo se anclan a átomos de azufre de cisteína del extremo C-terminal. Muchas proteínas que participan en el control de crecimiento y diferenciación celular se modifican así, incluyendo proteínas responsables del crecimiento descontrolado de tumores.
1. Un grupo prenilo (farnesilo (15C) o geranilgeranilo (20C)) se añade a una cisteína localizada 3 aminoácidos del extremo C-Terminal (secuencia XAAC)
2. Los aminoácidos anteriores a la cisteína son eliminados, dejando la cisteína en el extremo C-Terminal.
3. Un grupo metilo se añade al grupo carboxilo de la C-Terminal. Palmitoilación: el ácido palmítico (16C) se une a los átomos de azufre de
residuos internos de cisteína. o Cara extracelular:
“Anclas” de glicosilfosfatidilinositol (GPI): este anclaje se ensambla en la membrana del RE luego de completarse la síntesis. Glicolípidos que se unen a la secuencia hidrofóbica C-terminal ciertas proteínas. Las cadenas de ácidos grasos anclan el glicosilfosfatidilinositol a la membrana.
Regulación de la función proteica
Por pequeñas moléculas: la mayoría de enzimas se regulan por la unión de moléculas que inducen cambios en su conformación, provocando modificaciones en su actividad catalítica. Por ejemplo: la retroinhibición, que regula muchos factores de transcripción.
Intercambio GTP-GDP: las proteínas alternan entre las conformaciones de unión a GTP y de unión a GDP, activándose y desactivándose continuamente. Por ejemplo: la proteína Ras si se mantiene en su conformación activa unida a GTP, actúa como una señal contínua de división celular, promoviendo el crecimiento incontrolado de células tumorales.
Fosforilación de proteínas: es un proceso reversible en el que las enzimas proteína quinasas transfieren un grupo fosfato desde el ATP a un grupo hidroxilo de serina, treonina o tirosina (proteína-serina/treonina quinasas o proteína-tirosina quinasa). Dichas enzimas constituyen aproximadamente el 2% de los genes eucariotas. Este

Laura Mendoza~ 23 ~
proceso es revertido por las proteínas fosfatasas, que catalizan la hidrólisis de un grupo fosfato del aminoácido fosforilado.
Las proteínas quinasas pueden participar en vías de transducción de señales en la que una quinasa fosforila a una segunda quinasa que puede a su vez actuar sobre una tercera. Ésta acción secuencial puede transmitir una señal recibida en la superficie celular a una proteína diana en el interior de la célula, induciendo a cambios en el comportamiento celular en respuesta a estímulos ambientales. Por ejemplo: la degradación de glucógeno a glucosa-1-fosfato por acción de la epinefrina (adrenalina).
1. La epinefrina se une a su receptor en la superficie celular 2. Se convierte el ATP en AMPc, el cual se une a una proteína quinasa dependiente
de AMPc.3. La quinasa dependiente de AMPc fosforila una segunda proteína llamada
fosforilasa quinasa, la cual fosforila la enzima glucógeno fosforilasa.4. La glucógeno fosforilasa conduce a la degradación de glucógeno a glucosa-1-
fosfato.
Metilación y acetilación de histonas: para activar o reprimir la traducción. Nitrosilación: Adición de grupos NO a la cadena lateral de cisteína en una
modificación post-traduccional. Glicosilación en O de proteínas nucleares y citosólicas.
Interacciones proteína-proteína
Las proteínas están formadas por múltiples subunidades y la unión de una molécula reguladora altera la conformación de la proteína por cambios en las interacciones entre sus subunidades.
Por ejemplo, la proteína quinasa dependiente de AMPc está constituida por dos subunidades reguladoras y dos catalíticas en su conformación inactiva. Cuando se une el AMPc a las subunidades reguladoras se provoca un cambio conformacional que disocia el complejo, activando la proteína.
Degradación de proteínas
La cantidad de proteínas no solo está regulada por su tasa de síntesis son también por su tasa de degradación. La degradación se produce en respuesta a señales específicas, o cuando se reconoce una proteína dañada.
Vía de ubiquitina-proteasoma: es la ruta principal de degradación en eucariotas, usando la ubiquitina (76A) como un marcador de proteínas citosólicas o nucleares para una rápida proteólisis.
Ubiquitinación:1. La ubiquitina es activada por una enzima llamada enzima activadora de la
ubiquitina, E1, usando ATP.

Laura Mendoza~ 24 ~
2. La ubiquitina se transfiere a una segunda enzima, E2, llamada enzima conjugante de la ubiquitina.
3. La ubiquitina ligasa, E3, reconoce la proteína diana y se une a E2, transfiriendo la ubiquitina a la proteína.
Reconocimiento y degradación:4. Se une la ubiquitina al grupo amino lateral de una lisina.5. Moléculas de ubiquitina adicionales se añaden para formar una cadena de
varias ubiquitinas.6. Las proteínas poliubiquitinadas son reconocidas y degradadas (usando
ATP) por un complejo de múltiples subunidades llamado proteasoma. 7. Se libera la ubiquitina para que pueda reutilizarse.
La estabilidad de muchas proteínas se determina por su capacidad para ubiquitinarse.
La mayoría de las células contienen un único tipo de E1, varios tipos de E2 y varias familias de E3, pues reconocen a distintos sustratos de proteínas. Es la especificidad de estas enzimas lo que marca selectivamente las proteínas para la degradación.
La ubiquitina no solo interviene en la degradación de proteínas. La unión de moléculas sencillas de ubiquitina a algunas proteínas interviene en la regulación de la reparación y la transcripción del ADN y la endocitosis.
Las proteínas pueden modificarse por la unión de proteínas similares a la ubiquitina, por ejemplo SUMO, quienes actúan como marcadores de localización y regulación de la actividad proteica. Muchas proteínas modificadas con SUMO intervienen en el mantenimiento de la estructura de la cromatina.
Proteólisis lisosómica: La proteína entra en los lisosomas para ser digerida (autofagia)
Distribución y transporte de proteínas

Laura Mendoza~ 25 ~
La vía secretora es: RE – Golgi – vesículas de secreción/membrana plasmática/lisosomas/exterior de la célula.
Algunas proteínas pasan por las etapas iniciales de la vía secretora pero son retenidas en el R.E o en el aparato de Golgi.
Las proteínas destinadas a la vía secretora son transferidas al R.E mientras están siendo traducidas por los ribosomas unidos a la membrana (translocación cotraduccional), mientras que las proteínas destinadas al citosol, al nucleo, a mitocondrias, cloroplastos o peroxisomas son sintetizadas en ribosomas libres y posteriormente liberadas (translocación postraduccional), aunque toda la síntesis proteica se inicia en los ribosomas presentes en el citosol.
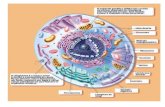
Retículo endoplasmático
Es una red de túbulos y sacos (cisternas) rodeados de membrana que se extiende desde la membrana nuclear por todo el citoplasma. Su membrana es aproximadamente la mitad de todas las membranas de la célula y la luz del retículo puede representar el 10% de todo el volumen celular.
R.E rugoso: cubierto por ribosomas, procesa proteínas. R.E de transición: desde donde parten vesículas hacia el aparato de Golgi.
Procesa proteínas. R.E liso: no está asociado a ribosomas y está implicado en el metabolismo de los
lípidos.
RE rugoso
Vía cotraduccional:
La marca que determina la unión del ribosoma con el R.E es una secuencia señal de aproximadamente 20 aminoácidos hidrófobos precedidos por un aminoácido básico en el N-terminal de la cadena polipeptídica creciente, que marca y dirige la cadena a los microsomas y que posteriormente es escindida por la peptidasa señal.
1. A medida que son sintetizadas, las secuencias señal son reconocidas y unidas a PRS, la cual está constituida por 6 polipéptidos y un ARN citoplásmico pequeño (ARNsrp). El ARNsrp se une tanto a las proteínas como al ARNr de la subunidad mayor del ribosoma.
2. La PRS inhibe la traducción y guía al complejo al R.E mediante la unión al receptor de la PRS en la membrana del RE. Éste receptor está formado por una subunidad α de 638 aminoácidos y una subunidad β de 300 aminoácidos, que son los sitios de unión a GTP.
3. Moléculas de GTP se unen tanto a PRS como al receptor, desencadenando la transferencia de la secuencia señal desde PRS al translocón. Estos translocones están constituidos por tres proteínas transmembrana llamadas proteínas sec61,

Laura Mendoza~ 26 ~
que es el núcleo central, receptor de ribosomas; y TRAMP que contacta la región N-terminal.
4. La hidrólisis de GTP libera a la PRS para ser reciclada.5. La transferencia del ribosoma-ARNm desde la PRS al translocón provoca que la
señal interaccione con cadenas laterales hidrofóbicas cortas del cuello del canal del translocón, transfiriéndose la cadena en crecimiento a través de él,
6. La peptidasa señal escinde la secuencia señal y el polipeptido es liberado en la luz del RER.
La translocación cotraduccional es dirigida por el procedimiento de la síntesis proteica.
Vía postraduccional:
Las proteínas sintetizadas en el citoplasma y dirigidas al RER luego de la traducción no requieren de una PRS. Sus secuencias señal son reconocidas por el complejo sec62/63 asociadas con el translocón de la membrana del RER.
Sin embargo, sí se necesitan de chaperonas citosólicas Hsp70 para mantener las cadenas en su conformación primaria para que puedan penetrar en el translocón y luego otras chaperonas Hsp70 dentro del RER (llamada BiP) necesaria para que atraviese el canal hasta el interior del RER. BiP dirige la translocación postraduccional.
Inserción de las proteínas en la membrana del RE
Las proteínas destinadas a incorporarse en la membrana plasmática o en la membrana del RE, Golgi o lisosomas, se insertan inicialmente en la membrana del RE, donde son transportadas como componentes de la membrana hasta su destino final por la misma vía de secreción.
Las proteínas integrales se incluyen en la membrana mediante regiones hidrofóbicas en hélice alfa de 20 a 25 aminoácidos que atraviesan la capa lipídica. La formación de hélices alfa maximiza los puentes de hidrógeno entre los enlaces peptídicos y las cadenas laterales hidrófobas interaccionan con las colas de ácidos grasos de los fosfolípidos.
Las proteínas pueden tener una o múltiples regiones que la atraviesan la membrana. Pueden tener el extremo N-terminal o el C-terminal hacia el citosol, y esta orientación se establece a medida que las proteínas en crecimiento se translocan en el RE. Los dominios proteicos expuestos a la superficie de la célula estarán en la luz del RE y Golgi.
Si se tienen aminoácidos positivos antes de la secuencia de pare la proteína tendrá el extremo n-terminal hacia el citosol. Si los aminoácidos positivos están luego de la secuencia de pare, el extremo N-terminal irá hacia el lumen.
Las proteínas que deben incorporarse a la membrana nuclear interna difunden lateralmente en la membrana del RE en lugar de transportarse en vesículas.
Un solo dominio trans-membrana:1. La secuencia señal N-terminal es escindida por la peptidasa señal.

Laura Mendoza~ 27 ~
2. Una segunda hélice alfa en el centro de la proteína que contiene una secuencia de detención de la transferencia, provoca el cierre del canal translocón, bloqueando que la cadena se siga translocando, por lo que el resto de la proteína debe sintetizarse en el citosol.
3. Se separan las subunidades del translocón y el dominio transmembrana penetra en la bicapa.
Varios dominios transmembrana: resultado de una serie alternante entre secuencias señal internas y secuencias transmembrana de detención de la transferencia.
Algunas proteínas también pueden anclarse a la membrana mediante secuencias señales internas que no son escindidas por la peptidasa señal, sino que son reconocidas por la PRS y trasladadas a la membrana por el mecanismo anterior.
Plegamiento y procesamiento
En el RE tiene lugar el plegamiento de las proteínas, el ensamblaje en varias subunidades, la formación de puentes disulfuro, las primeras etapas de la glicosilación y la adición de anclajes de glicolípidos.
Plegamiento: La chaperona BiP se une a la cadena sin plegar cuando cruza la membrana y después media el plegamiento y ensamblaje de las subunidades. Las proteínas correctamente ensambladas son liberadas de BiP y otras chaperonas y están disponibles para su transporte al aparato de Golgi. Las proteínas mal plegadas son dianas de degradación.
Formación de puentes disulfuro: El RE tiene un ambiente oxidante que junto a la disulfuro isomerasa media la formación de los puentes disulfuro.
Glicosilación. Anclaje de glicolípidos: se añade glicosilfosfatidilinositol a un residuo de
asparragina que ancla la proteína a la membrana. El complejo resultante es transportado a la superficie celular como componentes de la membrana.
Control de calidad:
Las chaperonas y las enzimas del procesamiento proteico en el lumen del RE actúan como sensores de proteínas mal plegadas. El proceso del control de calidad implica a BiP, otras chaperonas, la disulfuro isomerasa y otras proteínas accesorias.
1. Dos residuos de glucosa se escinden en la glicosilación, permitiendo la unión de la calreticulina, la cual es una proteína chaperona que facilita el plegamiento.
2. Se escinde el residuo de glucosa restante, liberando la calreticulina y la glicoproteína.
3. Un sensor de plegamiento dirige a las proteínas plegadas correctamente a hacia el RE transicional. Sin embargo, si el plegamiento es incorrecto, el sensor añade un nuevo residuo de glucosa para devolverla a la calreticulina, quien realiza una nueva tentativa de plegamiento.

Laura Mendoza~ 28 ~
4. Si el plegamiento es totalmente incorrecto, se envía a una vía de degradación mediante la retrotranslocación. En el citoplasma, la proteína se marca con ubiquitina y se degrada por el proteosoma.
Normalmente hay niveles suficientes de BiP no solo para plegar proteínas sino para unirse a moléculas señalizadoras y mantenerlas inactivas. Si hay un exceso de proteínas sin plegar (puede ser debido al estrés celular), estas compiten por el BiP, provocando la liberación de las moléculas que señalizan la respuesta a proteínas no plegadas, activándose la respuesta a proteínas no plegadas que consiste en:
Un aumento en la expresión de chaperonas La inhibición general de la síntesis proteica. Un aumento en la degradación de ARNm que codifica proteínas de la vía
secretora.Estas respuestas también inducen al aumento de la actividad del proteosoma y si estas medidas son insuficientes, se induce la muerte celular programada.
RE liso
Debido a su naturaleza hidrófoba, los lípidos se sintetizan asociados con membranas celulares ya existentes. Sin embargo, la mayoría se sintetizan en el RE y son transportados en vesículas o mediantes proteínas transportadoras.
El REL crece mientras más se consuma grasas o alcoholes (cirrosis) hasta que es demasiado grande.
La mayoría de los fosfolípidos derivan del glicerol y son sintetizados en la cara citoplasmática de la membrana del REL a partir de precursores hidrosolubles.
1. Dos ácidos grasos unidos a los transportadores de CoA se unen al glicerol-3-fosfato, liberándose los transportadores.
2. El fosfolípido resultante (ácido fosfatidico) se inserta en la membrana.3. Las enzimas de la cara citosólica del REL convierten el ácido fosfatidico en
diacilglicerol y catalizan la adición de diferentes grupos de cabezas polares, formando:
a. Fosfatidilcolinab. Fosfatidiletanolaminac. Fosfatidilserina: primero se añade etanolamina, luego ésta se sustituye por
serina.d. Fosfatidilinositol.
Los fosfolípidos nuevos solo se añaden a la mitad citosólica de la membrana del RE. Para mantener una membrana estable, los fosfolípidos deben transferirse a la mitad de la luz del RE. Esta transferencia requiere el paso de un grupo polar a través de la membrana, y es facilitada por unas flipasas, quienes catalizan la translocación de fosfolípidos y garantizan el crecimiento equilibrado de ambas mitades de la bicapa.
30-40% de la membrana.

Laura Mendoza~ 29 ~
El REL es responsable de la síntesis de los productos finales o de los precursores de los lípidos de membrana principales, por lo tanto se encuentra en grandes cantidades en células activas en el metabolismo lipídico, por ejemplo, en células que producen esteroides, como las de los testículos y del ovario. También es abundante en el hígado, donde tiene enzimas que metabolizan varios compuestos liposolubles, quienes inactivan un número importante de drogas potencialmente nocivas, como el fenobarbital, convirtiéndolas en compuestos hidrosolubles que son eliminados a través de la orina.
Exportación de lípidos y proteínas
Las vesículas que median el transporte de lípidos y proteínas a través de la vía secretora se originan por gemación en la membrana de un orgánulo y se funden con la membrana de otro.
Las proteínas deben estar marcadas por secuencias que señalizan su exporte o su retención en el RE. Muchas proteínas transmembrana tienen secuencias aminoacídicas diácidas o di-hidrofóbicas en sus dominios citosólicos que sirven como señales de exporte del RE. Estas proteínas transmembrana pueden actuar como receptores de proteínas ancladas por GPI y proteínas secretoras de la luz del RE.
Si se permite que las proteínas que funcionan en el interior del RE procedan a lo largo de la vía secretora, se perderán para la célula. Por lo tanto, muchas tienen una secuencia diana Lys-Asp-Glu-Leu (KDEL) en su extremo C-terminal que dirige su recuperación al RE. La retención de algunas proteínas transmembrana está dictada por secuencias C-terminales cortas con dos residuos de lisina (KKXX). Estas señales no impiden que las proteínas sean empaquetadas en vesículas y transportadas a Golgi, pero las proteínas con estas señales se unen a receptores específicos para el reciclado. Otras proteínas son recuperadas porque se unen a proteínas que tienen la secuencia KDEL, como BiP.
Aparato de Golgi
Está compuesto de cisternas (bolsas aplanadas rodeadas de membranas) y por vesículas asociadas. Funciona como una fábrica en el que las proteínas recibidas desde el RE se reprocesan y distribuyen para ser transportadas a su destino final. Y, además, los glicolípidos y la esfingomielina son sintetizados en el Golgi. En las células vegetales, Golgi es también el lugar donde se sintetizan los polisacáridos de la pared celular.
El aparato de Golgi es un orgánulo polarizado tanto en su estructura como en su función y contiene numerosos compartimentos, que se dividen en tres secciones: la red cis, el compartimiento medial y la red trans.
Todas las proteínas retenidas en el aparato de Golgi están asociadas con su membrana en lugar de ser solubles en su interior. Las señales responsables de esta retención se han localizado en sus dominios transmembrana, impidiendo que sean empaquetadas en vesículas y muchas veces las señales de las colas citoplasmáticas de algunas de estas proteínas son responsables de su recuperación desde compartimientos posteriores.

Laura Mendoza~ 30 ~
Las vesículas asociadas a Golgi no participan en el transporte de proteínas a través del aparato sino que devuelven a las proteínas residentes del Golgi a los compartimientos iniciales para su reutilización. Si se inhibe el transporte de vesículas desde el RE desaparece la estructura organizada del aparato de Golgi, la cual está basada en interacciones entre proteínas de las membranas de las cisternas y el citoesqueleto.
Proteínas
Las proteínas procedentes del RE entran por su cara cis, luego son transportadas a través del Golgi y salen por su cara trans. Según atraviesan el Golgi, las proteínas se modifican y distribuyen para el transporte a sus destinos finales. Algunas proteínas específicas se procesan en distintas regiones de cada cisterna.
Las proteínas del RE se transportan al compartimento intermedio RE-Golgi (CIREG o ERGIC) y pasan al aparato de Golgi a través de la red cis, en la que comienzan las reacciones de modificación. Luego atraviesan el compartimiento medial y el principio de la red trans donde son sometidos a nuevas modificaciones. El final de la red trans actúa como un centro de organización y distribución que dirige las moléculas hacia los endosomas, lisosomas, a la membrana o al exterior de la célula.
Glicosilación
La segunda parte de la modificación de N-oligosacáridos que se unieron a las proteínas en el RE tiene lugar en el aparato de Golgi:
Proteínas destinadas a ser secretadas o a la membrana plasmática
1. En la cisterna cis se eliminan cuatro residuos de manosa del extremo de la cadena del oligosacárido.
2. En la cisterna media se añade una N-acetilglucosamina a una de las manosas y se eliminan dos manosas más.
3. Se añade una fucosa (6C y un grupo aldehído) a una N-acetilglucosamina del extremo unido a la proteína y otras dos N-acetilglucosamina a la manosa libre.
4. En la cisterna trans se añaden tres galactosas a las N-acetilglucosamina de los extremos y tres residuos de ácido sialico (NANA) a las galactosas.
Las distintas glicoproteínas se modifican por mecanismos distintos y en grados diferentes dependiendo de la estructura de la proteína como de las enzimas presentes en los distintos compartimientos de Golgi de cada tipo celular implicadas en el proceso. Las glicosiltransferasas añaden azúcares, y las glicosidasas los eliminan.
Proteínas lisosómicas: la fosforilación ocurre en la red cis.
1. Se añade N-acetilglucosamina fosfato a residuos de manosa2. Se elimina un grupo N-acetilglucosamina, dejando residuos de manosa-6-fosfato.

Laura Mendoza~ 31 ~
Gracias a esto, los residuos de manosa no son eliminados durante el procesamiento posterior sino que es reconocido por un receptor de manosa-6-fosfato en la red trans que dirige el transporte a los lisosomas.
La enzima que añade la N-acetilglucosamina fosfato reconoce un determinante estructural específico presente en las proteínas lisosómicas, el cual no es simplemente una secuencia de aminoácidos sino que son regiones señal que dependen del plegamiento tridimensional de la proteína.
O-glicosilación: En el aparato de Golgi ocurre la adición secuencial de residuos únicos de azúcar. La serina o la treonina se unen directamente a la N-acetilgalactosamina, a la que se le pueden añadir otros azúcares, los cuales a veces sufren modificaciones por la adición de grupos sulfato en la red trans.
Distribución y exportación de proteínas
Las proteínas pueden ir desde Golgi hacia:
Un endosoma tardío y posteriormente a un lisosoma.
Las proteínas destinadas a la membrana lisosómica están marcadas por secuencias en sus colas citoplásmicas en vez de por residuos de manosa-6-fosfato.
Un endosoma de reciclaje. Vesículas secretoras. Directamente hacia la membrana plasmática: esto supone la incorporación de
nuevas proteínas y lípidos en la membrana y la secreción continua de proteínas celulares.
Las membranas de las células epiteliales se encuentran polarizadas, teniendo diferentes funciones entre sus dos dominios: el dominio apical y el dominio basolateral. Las proteínas que salen de la red trans se deben transportar selectivamente a dominios diferentes de la membrana polarizada. Esto se hace mediante el embalaje selectivo de proteínas en vesículas de transporte dirigidas a uno u otro dominio.
Síntesis de glicolípidos y esfingomielina
Esfingomielina: es el único fosfolípido no glicérico presente en las membranas (15%), sintetizada en la superficie luminal de Golgi por la transferencia de un grupo fosforilcolina desde la fosfatidil colina a la ceramida.
Glicolípidos: La adición de carbohidratos a la ceramida da lugar a diferentes glicolípidos. La glucosa se añade a la ceramida en la cara citosólica de Golgi. Sin embargo, la glucosilceramida se da la vuelta y los carbohidratos adicionales se añaden en la cara luminal de la membrana. Los glicolípidos no son capaces de translocarse a través de la membrana del Golgi.

Laura Mendoza~ 32 ~
Transporte de las vesículas
La selectividad del transporte es vital para mantener la organización funcional de la célula. La especificidad del transporte se basa en el empaquetamiento selectivo de la carga seleccionada en vesículas que reconozcan y se fusionen solo con la membrana diana apropiada.
Las vesículas de transporte que llevan proteínas secretoras están recubiertas con proteínas de la cubierta citosólica y por lo tanto se denominan vesículas cubiertas. La formación de estas vesículas está regulada por dos familias de proteínas pequeñas de unión a GTP: factores de ADP-ribosilación (ARFs 1-3 y Sar1) y muchas proteínas Rab.
Tres familias de proteínas de cubiertas de vesículas:
Clatrina: transporte bidireccional entre la red trans, los endosomas, los lisosomas y la membrana.
COPI: devuelven a las proteínas residentes a los compartimientos anteriores del RE o de Golgi.
COPII: transportan proteínas secretoras desde el RE al aparato de Golgi.
La formación de vesículas recubiertas con clatrina:
1. En la membrana, ARF/GDP es activada por ARF-GEF.2. ARF/GTP inicia el proceso reclutando una proteína adaptadora (GGA)3. Las proteínas adaptadoras reclutan un receptor transmembrana que tiene unida la
proteína a ser transportada4. GGA recluta otra proteína adaptadora, AP1, que sirve como sitio de ensamblaje
de la clatrina.5. La clatrina se ensambla en una estructura con forma de cesto que distorsiona la
membrana.
Durante el transporte, el GTP unido a ARF1 se hidroliza y el complejo ARF/GDP se recicla.
Fusión de vesículas:
La vesícula de transporte debe reconocer a la membrana diana correcta. Este reconocimiento está mediado por la interacción específica entre pares de proteína transmembrana, llamadas SNARE (v-SNARE y t-SNARE). El apareamiento de ambas proporciona la energía necesaria para acercar las bicapas lo suficiente como para desestabilizarlas y fusionarlas.
La llegada, unión y fusión de vesículas está mediada por complejos proteicos de ensamblaje secuencial (Rab). Las proteínas Rab individuales o combinaciones de ellas caracterizan distintos orgánulos y vesículas de transporte, otorgando especificidad.
1. Las proteínas Rab son transportadas a través del citosol unidas a GDP por inhibidores de la disociación de GDP (GDI).

Laura Mendoza~ 33 ~
2. En la membrana, son retirados por factores de desplazamiento de GDI y luego factores de intercambio de nucleótidos convierten Rab-GDP en Rab-GTP. Estos factores se localizan en membranas específicas y actúan sobre miembros específicos.
3. Rab-GTP recluta proteínas efectoras y a v-SNARE para ensamblar un complejo de pre-fusión. Sucede algo similar con t-SNARE.
4. Las proteínas efectoras unen las membranas mediante interacciones proteína-proteína.
5. Se estimula la hidrólisis de Rab-GTP y ambas SNARE se conectan enrollando sus hélices. Esto crea inestabilidad en las bicapas y se fusionan.
6. NSF/SNAP desensamblan el complejo SNARE hidrolizando ATP.
Lisosomas
Son el sistema digestivo de la célula. Tienen alrededor de 50 enzimas capaces de degradar todas las biomoléculas, bien sea material captado del exterior o componentes obsoletos de la misma célula.
En las levaduras y células vegetales, las vacuolas asumen la función de los lisosomas, además de almacenar nutrientes y mantener el equilibrio osmótico. Las proteínas destinadas a las vacuolas están marcadas por secuencias polipeptídicas cortas.
Las enzimas lisosomales son hidrolasas ácidas que actúan a pH ácido de aproximadamente 5. Esto proporciona una protección contra la digestión incontrolada, incluso si se rompiera la membrana lisosómica.
Para mantener el pH ácido los lisosomas deben concentrar activamente H+, mediante una bomba de protones que transporta protones desde el citosol hacia el lisosoma usando la energía de la hidrólisis del ATP, pues su concentración es aproximadamente 100 veces más elevada en el interior del lisosoma que en el citoplasma.
Formación del lisosoma
Se forman mediante la unión de vesículas de transporte de la red trans de Golgi con un endosoma tardío, que contiene las moléculas ingeridas por endocitosis.
Un cambio importante en la maduración de los endosomas es el descenso del pH interno que finalmente libera las hidrolasas ácidas desde la red trans de Golgi, dirigidas hacia los lisosomas por residuos de manosa-6-fosfato. Los endosomas tardíos se fusionan con lisosomas o maduran para convertirse en ellos.
Fagocitosis
Células especializadas (como los macrófagos) ingieren en fagosomas partículas grandes, incluyendo bacterias, restos de células y células envejecidas y se unen con lisosomas para degradarlas, formando fagolisosomas.

Laura Mendoza~ 34 ~
Autofagia
Es el recambio gradual de los componentes de la célula. Esta es una función de todas las células, está regulada en respuesta a la disponibilidad de nutrientes y es vital en el desarrollo embrionario, en la muerte celular programada y en la metamorfosis de insectos.
El primer paso es la formación de una envoltura de membrana citosólica alrededor de ua porción citoplasmática o un orgánulo. Posteriormente, la vesícula formada (un autofagosoma) se fusiona con un lisosoma, formando un fagolisosoma y se digiere el contenido.

Laura Mendoza~ 35 ~
Membrana plasmática
Todas las células están rodeadas por membrana, que define el límite de la célula y separa el contenido interno del medio externo. La membrana plasmática aísla al citoplasma y media las interacciones entre la célula y su medio, determinando la composición del citoplasma.
La estructura de los fosfolípidos es la responsable de la actuación de las membranas como barreras entre dos compartimientos acuosos.
La membrana es impermeable a moléculas hidrosolubles pues el interior de la bicapa está formada por cadenas de ácidos grasos.
Las bicapas son fluidos viscosos. Los ácidos grasos de la mayor parte de los fosfolípidos tienen enlaces dobles que
introducen codos en las cadenas y dificulta su empaquetamiento. Es ligera y flexible pues las largas cadenas hidrocarbonadas se mueven
libremente en el interior de la membrana. Los fosfolípidos y las proteínas son capaces de difundir lateralmente dentro de la
membrana.
Constitución de las membranas
Lípidos de membrana: elementos estructurales fundamentales.
Los fosfolípidos principales la fosfatidilcolina, fosfatidilserina, fosfatidiletanolamina y esfingomielina. Estos lípidos están distribuidos de manera asimétrica entre las dos mitades de la bicapa lipídica.
Fosfatidilcolina, esfingomielina → capa externa.Fosfatidiletanolamina, fosfatidilserina, fosfatidilinositol → capa interna. Las cabezas de la fosfatidilserina y del fosfatidilinositol están cargadas negativamente, por lo tanto, en la cara citosólica de la membrana hay una carga neta negativa.
Glicolípidos y colesterol
Los glicolípidos constituyen cerca del 2% de la membrana y están exclusivamente en la cara externa, con sus residuos hidrocarbonados expuestos a la superficie celular debido a su función de señalización.
El colesterol está distribuido más o menos igualitariamente en las dos mitades de la bicapa y más o menos en las mismas concentraciones molares que los fosfolípidos. Se inserta dentro de la bicapa con sus grupos polares hidroxilos cercanos a las cabezas de los fosfolípidos. Tiene la función de regular la fluidez de la membrana, a temperaturas muy altas el colesterol interfiere con el movimiento de las cadenas de ácidos grasos de los fosfolípidos, disminuyendo la fluidez de la parte externa y reduciendo su permeabilidad a moléculas pequeñas. A bajas temperaturas el colesterol interfiere entre las cadenas de ácidos, evitando que la membrana se congele. Las células bacterianas y vegetales carecen de colesterol.

Laura Mendoza~ 36 ~
Balsas lipídicas
El colesterol, los glicolípidos y la esfingomielina no difunden libremente sino que forman pequeñas placas semisólidas llamadas balsas lipídicas, las cuales se encuentran a un nivel superior de la mayoría de la bicapa pues las temperaturas de fusión de los esfingolipidos son distintas de las de los fosfolípidos derivados del glicerol. En estas balsas abundan las proteínas ancladas a GPI y otras proteínas que participan en la señalización celular y la endocitosis.
Pueden estabilizarse mediante interacciones con el citoesqueleto a través de proteínas periféricas de membrana, como caveolina. Varios tipos de proteínas entran y salen de las balsas, facilitando la agruàcoón de estas proteínas.
Proteínas de membrana: responsables de realizar las funciones específicas de la membrana.
Debido a que las proteínas son más grandes, hay aproximadamente una proteína por cada 50 o 100 lípidos, aunque las membranas plasmáticas están compuestas por un 50-50% de cada grupo.
Proteínas periféricas: Se asocian indirectamente a la membrana con interacciones proteína-proteína mediante enlaces iónicos, y se disocian de la membrana tras el tratamiento con agentes polares como soluciones de pH extremo o de alta concentración salina, que no rompen la bicapa.
Proteínas integrales: (transmembrana) Se insertan dentro de la bicapa, y solo pueden ser liberadas mediante tratamientos que rompan la bicapa alterando las interacciones hidrofóbicas, como los detergentes (moléculas anfipáticas).
La mayoría de las proteínas transmembrana son glicoproteínas con sus oligosacáridos expuestos en la superficie y pueden tener uno o varios dominios transmembrana, por ejemplo la banda 3, que consta de 929 aminoácidos y se cree que tiene 14 regiones que atraviesan la membrana y que forman dímeros que contienen canales internos a través de los cuales los iones pueden cruzar la membrana.
Las porinas, proteínas que forman canales en las membranas externas de algunas bacterias, no contienen regiones α hélices hidrofóbicas sino que atraviesan la membrana en forma de barriles β en los que 8 a 22 láminas β se pliegan para formar una estructura en forma de barril que encierra un poro acuoso donde las cadenas laterales de los aminoácidos polares revisten el poro y las cadenas de los aminoácidos hidrofóbicos interaccionan con los ácidos grasos.
Otras proteínas se unen a la capa interna de la membrana a través de lípidos asociados por enlace covalente. Estas proteínas son sintetizadas en ribosomas libres y son modificadas por la adición de lípidos (N-miristoilación, palmitoilación y prenilación), además de estar marcadas con secuencias positivas que interaccionan con los grupos cargados negativamente de la fosfatidilserina.

Laura Mendoza~ 37 ~
No todas las proteínas son capaces de difundir libremente por la membrana, en algunos casos, la movilidad está restringida por su asociación con el citoesqueleto, a otras proteínas de membrana, a proteínas de la superficie de células adyacentes o con la matriz extracelular. En las células polarizadas, la movilidad de las proteínas debe estar restringida a los dominios pertinentes de la superficie celular.
Glicocálix
Es un manto de carbohidratos que cubre a la célula, constituido por los oligosacáridos de los glicolípidos y las glicoproteínas, que sirve para:
Proteger la superficie celular frente al estrés iónico y mecánico. Crear una barrera ante organismos invasores. Marcadores de interacciones célula-célula.
Transporte de moléculas
Las proteínas de transporte específicas (carriers y proteínas de canal) son las responsables del tránsito selectivo de moléculas pequeñas a través de la membrana.
Simporte; transporte de dos moléculas en una misma dirección. Ejemplo: transporte activo de la glucosa.
Uniporte: transporte de una molécula. Antiporte: transporte de dos moléculas en direcciones opuestas. Ejemplo: antiporte
Na+-Ca+, Na+H+.
Potencial de membrana
La composición iónica del citoplasma es diferente a la del flujo extracelular. El potencial de membrana viene determinado por el flujo de todos los iones que la atraviesan y la apertura y el cierre de los canales iónicos vienen dados por la dinámica entre el potencial de reposo y el potencial de equilibrio de todos los iones de la célula.
Impulsos nerviosos
La llegada de un impulso nervioso al terminal de una neurona induce la liberación de neurotransmisores como la acetilcolina, estos neurotransmisores se unen a receptores en la membrana de células postsinápticas donde inducen la apertura de los canales iónicos regulados por voltaje.
Difusión pasiva
Es un proceso no selectivo en el que una molécula se disuelve en la bicapa fosfolipídica, difunde a través de ella y se disuelve en la solución acuosa al otro lado de la membrana. La dirección de este transporte viene determinada por las concentraciones relativas de la molécula dentro y fuera de la célula, a favor de un gradiente de concentración. Sólo las moléculas pequeñas e hidrófobas son capaces de transportarse por este método (O2, CO2, etanol, H2O)

Laura Mendoza~ 38 ~
Difusión facilitada
Viene determinada por sus concentraciones relativas dentro y fuera de la célula (en el caso de las moléculas cargadas, por su potencial eléctrico) y no interviene ninguna fuente de energía, sin embargo, las moléculas no se disuelven en la bicapa sino que su tránsito viene mediado por proteínas que permiten a las moléculas atravesar la membrana sin interaccionar con su interior hidrofóbico.
Proteínas transportadoras: se unen en un lado de la membrana a las moléculas que han de ser transportadas, sufren un cambio conformacional que libera a la molécula del otro lado (azúcares, aminoácidos, nucleósidos)
El transportador de glucosa presenta 12 segmentos transmembrana en α hélice formada por aminoácidos hidrófobos y ciertos aminoácidos polares que forman el sitio de unión de la glucosa. Este transporte es reversible y ocurre principalmente en las células hepáticas.
Proteínas de canal: forman poros abiertos a través de la membrana y permiten la difusión de moléculas con el tamaño y la carga apropiados. Por ejemplo, las acuaporinas, que permiten el paso de H2O más rápidamente que por difusión pasiva, y que mantienen el equilibrio hídrico en el cerebro y expulsan el sudor de la piel.
Canales iónicos
Su apertura y cierre es extremadamente rápido (un millón de iones por seg). Altamente selectivos gracias a que tienen un tamaño específico. No se encuentran permanentemente abiertos, su apertura está regulada por
puertas que se abren en respuesta a estímulos específicos.
La apertura y cierre de estos canales tiene una función importante en células nerviosas y musculares pues permiten la transmisión de señales eléctricas.
Los canales regulados por ligando se abren en respuesta a neurotransmisores u otras moléculas señal.
En su estado cerrado, el poro de un canal puede estar bloqueado por las cadenas laterales de los aminoácidos hidrofóbicos, cuando se une la proteína a ser transportada se induce un cambio conformacional y las cadenas laterales hidrofóbicas dejan de estar en el canal, permitiendo el paso de moléculas con carga positiva pero no negativa, pues el canal está recubierto de aminoácidos cargados negativamente.
Los canales regulados por voltaje se abren en respuesta a variaciones del potencial eléctrico.
Los canales regulados por voltaje presentan un mayor grado de selectividad iónica.
Canales para Na+ (+H2O): viene dada por la presencia de un poro estrecho que actúa como filtro de tamaño.

Laura Mendoza~ 39 ~
Los canales de Na+ y de Ca+ están formados por una cadena polipeptídica que contiene cuatro dominios repetidos que corresponden a la subunidad del canal K+. El dominio cuatro es el que regula la apertura por voltaje pues contiene múltiples aminoácidos positivos que se desplazan en respuesta a cambios en el potencial.
Canales para K+: está formado por cuatro subunidades idénticas, con varias hélices transmembrana. Forman canales estrechos, pero para impedir el paso de moléculas más pequeñas usa un mecanismo que consiste en un filtro estrecho que está delimitado por oxígenos carbonílicos (C=O). Cuando un ión K+ entra en el filtro, interacciona con estos oxígenos carbonílicos y las moléculas unidas a K+ se desplazan, dejando al K+ deshidratado pasar a través del poro. Un Na+
deshidratado es muy pequeño para interaccionar con el filtro selectivo.
Transporte activo
Se usa la energía de la hidrólisis de ATP para dirigir el transporte de las moléculas en dirección energéticamente desfavorable.
Bombas iónicas: son las responsables de mantener el gradiente iónico a través de la membrana plasmática. Estas bombas son saturables.
Bomba de Na+-K+: Está compuesta por 4 polipéptidos, 2α (grandes, unión de Na+) y 2β (pequeñas) y usa del 30-50% de ATP. La concentración de Na+ en la célula es cerca de diez veces superior fuera que dentro de la célula y la de K+ es mayor dentro que fuera. Es vital para la propagación de señales eléctricas en el nervio y el músculo, para mantener el equilibrio osmótico y el volumen celular, pues el citoplasma contiene alta concentración de moléculas orgánicas que en ausencia de algo que lo compense, provocaría un flujo de agua hacia el interior celular por ósmosis.
1. 3 Na+ se unen a sitios expuestos dentro de la célula.2. La unión estimula la hidrólisis de ATP, fosforilando la bomba.3. El Na+ se libera al exterior de la célula.4. Al mismo tiempo, dos K+ se unen a sitios expuestos en la superficie celular5. Se desfosforila la bomba.6. Se libera el K+ al interior de la célula.
El potencial de membrana creado por el flujo de K+ dirige al Cl- fuera de la célula, donde la concentración es mayor que en el citoplasma.
Bomba de Ca+: transportan iones de calcio al exterior celular o a la luz del RE por lo que las concentraciones intracelulares son extremadamente bajas, provocando que la célula sea sensible a pequeños incrementos del nivel de Ca+, el cual desempeña un papel en la señalización celular.

Laura Mendoza~ 40 ~
Transporte de H+
El H+ es bombeado hacia fuera de las células que recubren el estómago, provocando la acidez del jugo gástrico. El transporte activo de H+ hacia los lisosomas y endosomas son bombas estructuralmente distintas. También existe una ATP sintetasa en mitocondrias y cloroplastos, donde la bomba opera en sentido contrario, usando el movimiento de los iones en contra del gradiente para dirigir la síntesis de ATP.
Transportadores ABC
Son transportadores de membrana caracterizados por dominios de unión a ATP. El ser humano tiene al menos 48 genes que codifican transportadoras.
En bacterias, introducen una amplia gama de nutrientes y en células eucariotas transportan sustancias tóxicas al exterior.
Están relacionados con la resistencia a ciertos fármacos de quimioterapia de células cancerosas y con la fibrosis quística, en donde el producto del gen responsable de la enfermedad (CFTR) es un canal de Cl- de transporte defectuoso en células epiteliales.
Transporte activo dirigido por gradientes iónicos
Es el transporte en contra del gradiente de concentración empleando la energía derivada de acoplar el transporte de una segunda molécula en la dirección favorable energéticamente. Por ejemplo, la bomba Na+-K+ proporciona una fuente de energía empleada para alimentar el transporte activo de azúcares, aminoácidos e iones.
La toma de glucosa en células intestinales se hace mediante un transporte de dos iones de Na+ y una glucosa hacia el interior celular.
Endocitosis
Es la captación de macromoléculas, donde el material a ser inducido es rodeado por una porción de la membrana que luego se invagina para formar una vesícula.
La endocitosis mediada por receptor es un mecanismo para la entrada selectiva de macromoléculas, quienes se unen a receptores de la superficie celular, los cuales están acumulados en regiones especializadas llamadas depresiones revestidas con clatrina, que tienen una vida media de aproximadamente 2 minutos. La endocitosis mediada por receptor provoca la entrada no selectiva de fluido extracelular.
La dinamina, una proteína de unión a GTP, se dispone en anillos alrededor del cuello de estas depresiones revestidas con clatrina y las invagina para formar vesículas revestidas con clatrina, quienes se fusionan con endosomas tempranos y su contenido se distribuye.
El colesterol se transporte a través del torrente sanguíneo en forma de partículas lipoproteínicas como LDL, la cual necesita de receptores específicos para entrar en la célula. Las personas que padecen de hipercolesterolemia familiar son incapaces de introducir LDL desde los fluidos extracelulares, bien sea por la ausencia de un receptor,

Laura Mendoza~ 41 ~
como por el hecho de que no estén acumulados en las depresiones revestidas. Estas mutaciones pueden ser tan simples como un cambio de aminoácidos.
Las señales de internalización de muchos receptores están compuestas por seis aminoácidos en los que la tirosina es esencial. Un receptor de LDL hace un ciclo de ida y vuelta en aproximadamente 10 minutos, algunos receptores son degradados en los lisosomas.
Las caveolas son pequeñas invaginaciones de la membrana plasmática organizadas por la acción de la caveolina. Los lípidos de las caveolas e incluso la caveolina pueden mediar como receptores para macromoléculas como la HDL.
La macropinocitosis es la internalización de fluidos mediante vesículas grandes.

Laura Mendoza~ 42 ~
Mitocondrias
Son los orgánulos responsables de generar la mayoría de la energía útil derivada de la degradación de los lípidos y carbohidratos, que es convertida en ATP mediante la fosforilación oxidativa.
La oxidación del acetil CoA a CO2 está acoplada a la reducción de NAD+ y FAD a NADH y FADH2, los cuales se transfieren al O2 a través de una serie de transportadores de la membrana. La energía de estas reacciones se convierte en energía potencial acumulada como un gradiente de protones a través de la membrana, que se usa para dirigir la síntesis de ATP.
Contienen varias copias por orgánulo de su propio genoma, distinto al código universal, formado por moléculas circulares de ADN que contiene:
13 secuencias codificadoras de proteínas, que son designadas como componentes de los complejos respiratorios I, III, IV o V.
Intrones. Genes para los ARNr 12 y 16S (23S, 16S y 5S en bacterias y plantas) y
para 22 ARNt, en los cuales la U en la tercera posición del anticodón se puede aparear con cualquiera de las cuatro bases.
Una región llamada bucle D que contiene un origen de replicación del ADN y secuencias promotoras de la transcripción.
No son orgánulos estáticos, sino que se fusionan continuamente entre si y se dividen en dos, que es posible que permita el intercambio de material genético.
Casi todas las mitocondrias (incluyendo mutaciones en el orgánulo) son heredadas de la madre pues se necesita mucha energía para mover los espermatozoides, por lo que las mitocondrias se ubican en el cuello (flagelo), el cual se desprende al ser fecundado. Las mutaciones pueden estar relacionadas con el síndrome metabólico (obesidad y diabetes), con neuropatías ópticas, etc. La acumulación progresiva de mutaciones en el ADN mitocondrial durante la vida de los individuos contribuye al envejecimiento.
Las mitocondrias de tejidos diferentes contienen proteínas distintas. Su cantidad depende de la función de la célula hay 80% en neuronas, 60% en
células musculares y 40% en células miocárdicas Están rodeadas por un sistema de doble membrana, una interna y la otra externa.
La membrana interna forma numerosos pliegues (crestas) que se extienden hacia la matriz mitocondrial. La membrana interna es el lugar principal de generación de ATP y eso se ve reflejado en
El incremento de su superficie mediante el plegamiento en crestas. Está compuesta en un 70% de proteínas, que intervienen en la fosforilación
oxidativa y en el transporte de metabolitos entre el citosol y la mitocondria. Es impermeable a la mayoría de los iones y las moléculas pequeñas, lo cual es
vital para mantener el gradiente de protones.

Laura Mendoza~ 43 ~
La membrana externa es completamente permeable a moléculas pequeñas, ya que contiene porinas que forman canales que permiten la difusión libre de moléculas menores de 1000 daltons.
La matriz contiene el sistema genético mitocondrial y las enzimas responsables de las reacciones del metabolismo oxidativo. Las etapas iniciales de la glucólisis tienen lugar en el citoplasma, donde la glucosa es transformada en piruvato. El piruvato es transportado al interior de la mitocondria, donde se oxida a acetil CoA, la cual es degradada a CO2
produciendo ATP. Los ácidos grasos también producen acetil CoA.
Internalización de proteínas
A pesar de tener su propio genoma, las proteínas requeridas para la replicación, transcripción o traducción del ADN, así como los genes que codifican la mayoría de las proteínas mitocondriales requeridas para la fosforilación oxidativa y las enzimas que intervienen en el metabolismo mitocondrial, no están en las mitocondrias sino en el núcleo.
La mayoría de las proteínas son marcadas y dirigidas a la mitocondria mediante secuencias N-terminales de 20 a 25 aminoácidos (presecuencias) con múltiples residuos de aminoácidos con carga positiva en forma de hélice α que se escinden tras entrar en el orgánulo.
1. Las presecuencias se unen a receptores en la superficie de las mitocondrias, Tom22, Tom20 y Tom5
2. La cadena se inserta en un complejo proteínico (complejo Tom), que contiene una Tom40, que dirige la translocación a través de la membrana externa.
3. Las proteínas son transferidas a un segundo complejo en la membrana interna (complejo Tim). La mayoría de las proteínas con presecuencias atraviesan la membrana interna a través del complejo Tim23, aunque algunas tienen una segunda cadena hidrofóbica, salen lateralmente de éste complejo para insertarse en la membrana.
Debido a la transferencia de protones desde la matriz al espacio intermembrana se establece un potencial eléctrico, siendo la matriz negativa. Esto dirige la translocación de la presecuencia cargada positivamente.
Las proteínas deben estar al menos parcialmente desplegadas, por lo que se requieren chaperonas Hsp70 en el lado citoplasmático, otra Hsp70 asociado con el complejo Tim23 que actúa como un motor que emplea la hidrolisis del ATP para internalizar las proteínas.
4. La presecuencia es entonces escindida por una peptidasa procesadora de la matriz (MPP) y la proteína se une (usando ATP) a otras chaperonas Hsp70 de la matriz para ser plegadas. Algunas se transfieren a una chaperonina donde se producen plegamientos adicionales.

Laura Mendoza~ 44 ~
Distribución de proteínas
Membrana interna: No tienen presecuencias, sino múltiples secuencias de internalización, por lo tanto, no son reconocidas por Tim20. Estas proteínas, en asociación con una chaperona Hsp90 son reconocidas por un Tom70 y translocadas mediante Tom40.
Las proteínas de la membrana interna son transportadoras de moléculas pequeñas (proteínas transmembrana multipase) que intercambian nucleótidos e iones entre la mitocondria y el citosol.
Intermembrana: son reconocidas por el complejo Tim22. Proteínas Tiny Tims actúan como chaperonas y transportadoras que escoltan proteínas hasta Tim22. Las proteínas son parcialmente translocadas hasta que señales internas de detención de la transferencia causen su salida lateral del poro.
Proteínas destinadas a la membrana externa, interna o al espacio intermembrana tienen tanto una presecuencia como una secuencia señal interna.
Proteínas con dominios transmembrana: salen lateralmente del canal Tom40. Proteínas intermembrana: pasan a través de la membrana externa pero son
reconocidas por chaperonas específicas para cisteína, reteniéndolas en el espacio intermembrana.
Otras son transportadas a la matriz a través del complejo Tim23 y al eliminarse la presecuencia se deja expuesta una señal que dirige a la proteína a través de un complejo Oxa1, donde pasan al espacio intermembrana o son insertadas en la membrana interna.
Proteínas de la membrana externa: como Tom40 y porinas (barril β) salen hacia el espacio intermembrana a través del complejo Tom, donde son reconocidas por las tiny tims y transportadas al complejo SAM (maquinaria de distribución y ensamblaje) desde donde se insertan en la membrana interna.
Los fosfolípidos también son importados desde el citosol. La fosfatidilcolina y la fosfatidiletanolamina se transportan a las mitocondrias mediante proteínas de transferencia de fosfolípidos, las cuales protegen el sitio de unión hidrofóbico de la proteína.
Las mitocondrias sintetizan fosfatidilserina a partir de fosfatidiletaloamina y catalizan la síntesis de la cardiolipina.
Fosforilación oxidativa
Produce la mayor parte de la energía utilizable obtenida de la degradación de los carbohidratos o los ácidos grasos, tiene lugar en el interior de las mitocondrias.
La glucolisis y el ácido cítrico producen en total cuatro moléculas de ATP, diez NADH y dos FADH2. Los electrones del NADH y el FADH2 son transferidos al O2 mediante la fosforilación oxidativa, formando de 32 a 34 moléculas de ATP.

Laura Mendoza~ 45 ~
Cadena de transporte de electrones
La trasferencia de electrones desde el NADH al O2 es una reacción que desprende mucha energía (-52,5kcal/mol/2e-). Esto debe hacerse paulatinamente a través de cuatro complejos transportadores que constituyen la cadena de transporte de electrones o una gran parte se perdería en calor. Un quinto complejo acopla las reacciones del transporte de electrones a la síntesis de ATP.
1. Los electrones del NADH entran en la cadena de transporte en el complejo I.2. Los electrones se transfieren del NADH a un nucleótido de flavina y luego, a través
de un transportador de hierro-azufre, a la coenzima Q, que lleva los electrones hasta el complejo III. Q es una molécula pequeña, liposoluble.
3. Los electrones se transfieren del citocromo b al citocromo c, una proteína periférica de membrana unida a la cara externa de la membrana interna, que transporta los electrones al complejo IV.
4. El complejo IV transfiere los electrones al O2 5. El complejo V acopla el transporte de electrones a la síntesis de ATP.
El complejo II recibe los electrones del succinato del ciclo de Krebs y los transfiere a FADH2
Acoplamiento quimiosmótico
La energía derivada del transporte de electrones está acoplada a la formación de un gradiente de protones en la membrana mitocondrial interna pues en cada complejo se transportan 4 protones por cada par de electrones.
El gradiente de protones a través de la membrana interna corresponde a una concentración de protones diez veces menor en la matriz, que tiene un pH de 8, mientras que el espacio intermembrana tiene un pH de 7. Este gradiente genera un potencial eléctrico de 0,14V, siendo la matriz negativa.
El complejo V o ATP sintetasa está constituida por F0 y F1, los cuales están unidos por un tallo estrecho. F0 es un motor de energía eléctrica que atraviesa la membrana formando un canal. La subunidad F1 cataliza la síntesis de ATP a partir de ADP e iones fosfato (F i) mediante el retorno de protones a la matriz.
Se requiere el flujo de 4 protones a través de F0 para sintetizar una molécula de ATP.
Transporte de metabolitos
Está mediado por proteínas transportadoras y dirigido por el gradiente electroquímico.
El ATP es exportado mediante un intercambio de ATP por ADP dirigido por el voltaje. El transporte de Pi y de piruvato se hace intercambiando alguno por OH- y es el pH el que dirige el intercambio.
Peroxisomas

Laura Mendoza~ 46 ~
Contienen al menos 50 enzimas que intervienen en diversas rutas metabólicas, incluyendo la degradación de ácidos grasos (proceso exclusivo de los peroxisomas en levaduras y plantas mas no en humanos) y el metabolismo de la fotosíntesis. La mayoría de las células humanas contienen unos 500 peroxisomas, los cuales se multiplican mediante división y pueden ser regenerados.
Sus proteínas, (pex1, pex2) se codifica el genoma nuclear y son sintetizadas por ribosomas libres e importadas al orgánulo. Entre ellas, encontramos a la catalasa, que descompone el peróxido de hidrógeno, el cual es nocivo para la célula, en agua o lo utiliza para degradar otro compuesto. Por lo tanto, los peroxisomas actúan como un compartimiento para las reacciones de oxidación.
Los peroxisomas también sintetizan dolicol, colesterol y plasmalógenos. En el hígado, intervienen en la síntesis de ácidos biliares. Realizan diferentes reacciones bioquímicas en distintos tejidos.
Formación
Pex3, una proteína transmembrana ubicada junto a pex9 en el RE, recluta a pex19, una proteína farnesilada del citosol, en el RE, las vesículas que contienen estas proteínas se separan y se fusionan con peroxisomas preexistentes o entre ellas.
Las proteínas actúan como receptores de importe para otras peroxinas mediante una secuencia ser-lys-leu en el extremo C-terminal (PTS1) o una secuencia de 9 aminoácidos en el extremo N-terminal (PTS2). Estas secuencias por lo general no se escinden.
Las proteínas destinadas a las mitocondrias, cloroplastos y peroxisomas se sintetizan en los ribosomas libres y son importadas a sus orgánulos de destino como cadenas polipeptídicas completas.
Su proceso de maduración implica el importe de diferentes clases de proteínas desde el citosol.

Laura Mendoza~ 47 ~
Diferencias entre la transcripción en procariotas y eucariotas
Característica Procariota Eucariota
Promotor Cajas y zona operadora Solo cajas
Cistrón Policistrones Monocistrones
ARN polimerasa Una con 5 subunidades Tres ARN polimerasas con 13, 12 y 17 subunidades respectivamente
Estabilización No tiene Al comienzo de la cadena, 7-metil-guanosina (CAP) y al final, una cola poliA.
Comienzo La ARN pol se autoacopla al promotor
ARN pol necesita la presencia de factores de transcripción
Intrones No tiene Tiene y se eliminan mediante splicing
Lugar de acción Inmediatamente En el citoplasma
Conformación de la PRS
ARNsrp (300 nucleótidos) Se une tanto a las proteínas como al ARNr de la subunidad mayor del ribosoma.
P9 y P14 Detiene el alargamiento de la cadenaP54 Se une a la secuencia señal y une al
GTP.P68 y P72 Translocación a la membranaP19 Enlaces entre P54 y un extremo de la
cadena de ARNsrp

Laura Mendoza~ 48 ~
Toms y Tims
Nombre FunciónTom20 Reconoce la presecuenciaTom70 Reconocer proteínas de membrana
internaTom40 Translocar proteínas desde el citosol al
espacio intermembranaTim 22 Reconocer proteínas intermembranaTiny tims Dirigir a las proteínas hacia Tim22 o a
SAMTim 23 Transportar proteínas desde la
membrana interna a la matriz mitocondrial
Complejos de la cadena transportadora de electrones
Nombre Composición Energía liberada
Complejo I 40 polipéptidos -16,6kcal/mol
Complejo II 4 polipéptidos
Complejo III 10 polipéptidos -10,1kcal/mol
Complejo IV -25,8kcal/mol
Complejo V F0 y F1 1ATP/4H
Términos de interés

Laura Mendoza~ 49 ~
AMPc: adenosin monofosfato cíclicoARN: ARN FunciónMensajero (ARNm) Molde para la síntesis de proteínasTransferencia (ARNt) Traducción del ARNRibosómico (ARNr) Traducción del ARNARN no codificante (MicroARN) Corte y empalme de ARNm.
Clasificación de proteínas en eucariotas.Reguladores de la expresión génica y de la traducción.
ARNsi: short interfering ARN.ARF: ADP-ribosylation factor. BRE: B recognition element; secuencia reconocida por TFIIB.Cistrones: Conjunto de varios codones Segmento de ADN compuesto de intrones y exones.
Monocistrónico: Información genética solo para una cadena polipeptídicaPolicistrónico: Información genética para varias cadenas polipeptídicas de
función relacionada, propio de procariotas.Codón: unidad del código genético compuesta de 3 pares de bases de ADN que codifican para un aminoácido.Corriente abajo: Va desde el lugar de inicio de la transcripción (+1) hacia delante.Corriente arriba: Va desde el lugar de inicio de la transcripción (+1) hacia atrás.DPE: Downstream promoter element; secuencia reconocida por TFIIDDCE: Downstream core element; reconocido por TFIID.DSIF: DRB sensitivity inducing factor.Fagocitosis: función principal de dos glóbulos blancos sanguíneos: macrófagos y neutrófilos (fagocitos profesionales), quienes desempeñan un papel crítico eliminando los microorganismos de los tejidos infectados.Gemación: Reproducción asexual. Una división desigual donde se forman prominencias sobre el individuo progenitor, y que al crecer y desarrollarse origina nuevos seres que pueden separarse del organismo parental o quedar unidos a él.Gen: secuencia de nucleótidos en la molécula de ADN equivalente a una unidad de transcripción. Contiene la información que sintetiza para un polipéptido, una enzima o un ácido ribonucleico (ARNm, ARNt, ARNr)Glicofosfatidilinositol: glicolípido (oligosacárido unido a un lípido) cuyo oligosacárido constituyente está formado por glucosamina y manosa unidos al residuo inositol del lípido, con un extremo etanolamina que sirve de puente para el extremo C-Terminal de una proteína. HDL: high density lipoprotein.HGM: High movility group. INR: secuencia iniciadora, se une al factor TFIID y a la polimerasa II.LDL: low density lipoprotein.MPP: matrix processing peptidase.

Laura Mendoza~ 50 ~
MTE: Motif ten element; ubicado de 18-27 nucleótidos corriente abajo. La pérdida de actividad de transcripción de la caja TATA o de DPE por mutaciones puede ser compensada por la adición de una secuencia MTE.NELF: Negative elongation factor. NTP: ribonucleósidos 5’-trifosfato.PABP: proteína de unión a poli-A (poly-A binding protein)Plasmalógenos: fosfolípidos en los que una de las cadenas carbonadas está unida no mediante enlace éster sino por un enlace éter. Son componentes importantes de la membrana de tejidos como el cerebro y el corazón.Polimerización: proceso químico en el cual se unen varias moléculas de un compuesto para obtener una macromolécula. PRS: partícula de reconocimiento de la señalRISC: RNA induced silencing complexRNPsn: (small nuclear ribonucleoprotein particles) partículas pequeñas de rinonucleoproteínas nuclearesSL1: factor de selectividad 1. Se compone de cuatro subunidades proteínicas, una de las cuales es TBP.SNARE: Soluble NSF Attachment Protein Receptor.SUMO: small ubiquitin-related modifier.Tándem: repetición de la misma secuencia una y otra vez, todas seguidas.UBF: (upstream binding factor) factor de unión corriente-arriba.